آموزش طراحی پرایمر برای ریل تایم ( Real Time PCR)
آموزش مقدماتی ریل تایم پی سی آر (Real Time PCR) و کاربرد رنگ سایبر گرین (SYBR Green)
Real Time PCR بسیار شبیه به واکنش PCRمی باشد اما تفاوت آن با PCR معمولی به کار گرفتن یک نشانگر فلورسنت در واکنش جهت ردیابی تکثیر محصول واکنش می باشد. این گزارشگرها به گونه ای طراحی میشوند که در صورت تکثیر DNA تولید نور کنند بنابراین نور بیشتر برابر است با تکثیر محصول و افزایش شدت نور ثبت شده در دستگاه با میزان محصول بدست آمده نسبت مستقیم دارد.
یکی از این نشانگرهای فلورسنت، رنگ سایبرگرین (SYBR® Green) و یا اوا گرین (Eva green) می باشند. دراینجا از رنگ های باند شده به DNA دو رشته ای به عنوان گزارشگر فلوروسنت برای مشاهده واکنش PCR استفاده می شود. این دو رنگ به شکاف کوچک مارپیچ دو رشته ای DNA باند می شود و سبب ایجاد نور فلورسنت می گردند، اما رنگ هایی که به رشته DNA باند نشده اند فلوروسنت خیلی کمی را نشان می دهند (شکل 1).
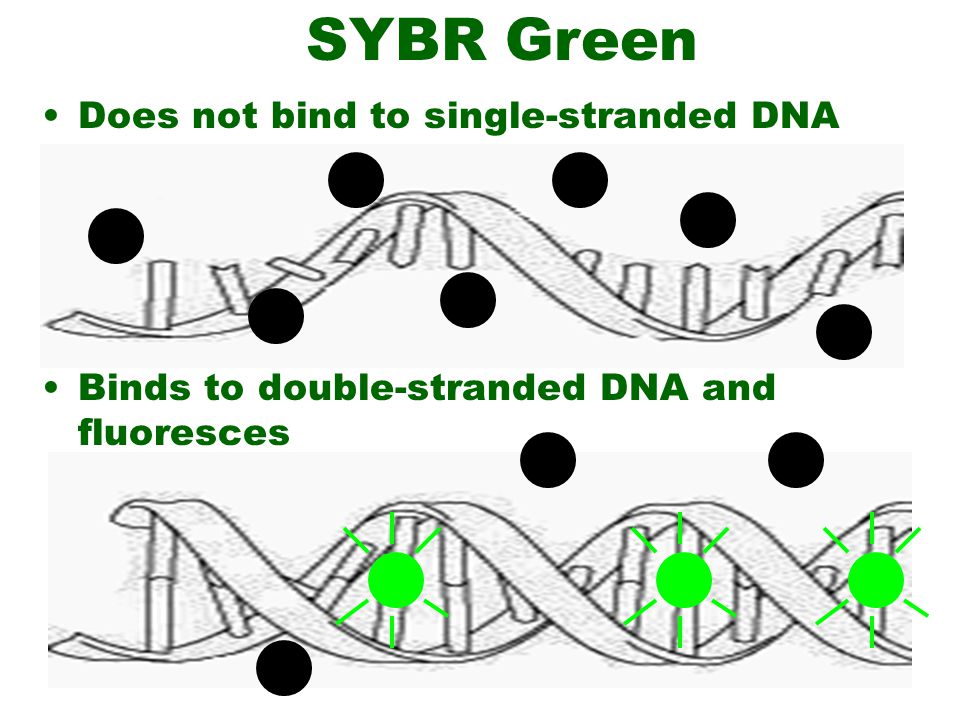
شکل 1: این شکل بیانگر قابلیت اتصال رنگ های فلورسنت به DNA می باشد.
بنابراین در روش ریل تایم، با تکثیر DNA قطعات دو رشته ای افزایش می یابند و با اتصال رنگ های فلورسنت به آن ها میزان نور انتشار شده نیز بیشتر می گردد. و این انتشار نور فلوروسنت در سیکل های متوالی بر اثر مضاعف شدن محصول PCR افزایش می یابد (شکل 2) بنابراین با ثبت مقدار فلوروسنت ساطع شده در هر سیکل، می توان پیشرفت تکثیر محصولات واکنش PCR را در طول مرحله نمایی PCR مشاهده نمود.
نکته: رنگ های سایبر گرین (SYBR® Green) و یا اوا گرین (Eva green) قابلیت اتصال به DNA تک رشته را ندارند.
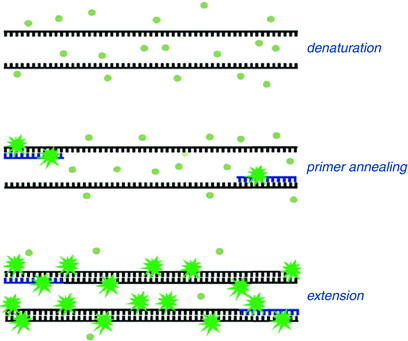
شکل 2: عملکرد رنگ های فلورسنت را در اتصال به DNA دورشته ای در روش ریل تایم.
آموزش اختصاصی طراحی پرایمر برای ریل تایم با روش سایبر گرین ( Real Time PCR SYBR Green)
در روش ریل تایم نیز مانند روش PCR ما نیازمند طراحی پرایمر می باشیم. برای طراحی پرایمر در روش ریل تایم علاوه بر رعایت نکات عمومی در طراحی پرایمر (که در ادامه به آن اشاره می نماییم) باید به یک سری نکته اختصاصی در طراحی پرایمر توجه نمود که در ادامه به این نکات اشاره می نماییم.
نکته اول در طراحی پرایمر در بررسی بیان ژن ها: پرایمرهای طراحی شده نباید بر روی توالی ژنومی متصل و محصولی را تکثیر نمایند.
در بررسی بیان ژن ها ابتدا از نمونه ها، RNA استخراج شده و سپس RNA استخراج شده توسط آنزیم reverse transcriptase (آنزیم RT) به cDNA تبدیل می گردد. سپس بررسی بیان ژن ها با تعیین میزان نسخه های رونویسی شده از هر ژن (mRNA) در cDNA سنتز شده، صورت می پذیرد. بنابراین پرایمرها باید برروی توالی mRNA ژن ها طراحی شود و در واکنش باید از cDNA به عنوان الگو استفاده شود. اما از آن جاییکه در بیشتر موارد RNA استخراج شده و cDNA سنتزشده به DNA ژنومی آلوده است بنابراین طراحی پرایمرها باید به صورتی باشد که یا به توالی DNA ژنومی متصل نشود و یا در صورت اتصال به DNA، محصولی از الگوی DNA تکثیر نشود.
البته در موقع سنتز cDNA می توان از آنزیم DNase جهت حذف آلودگی DNA استفاده نمود، اما استفاده از آنزیم DNase نیز مشکلات بسیاری را ایجاد می کند. مشکل اول این است که آنزیم DNase سبب کاهش کیفیت RNAها و کاهش راندمان سنتز cDNA می گردد. بنابراین ممکن است بعد از سنتز cDNA تکثیر قطعه مورد نظر به خوبی صورت نگیرد.
مشکل دوم استفاده از آنزیم DNase این است که جهت استفاده از این آنزیم به میزان بالایی از RNA نیاز است. بنابراین هنگامی که میزان RNA در دسترس کم باشد، در این شرایط تیمار نمونه های RNA با آنزیم DNase سبب عدم سنتز cDNA می گردد.
مشکل سوم استفاده از آنزیم DNase این است که استفاده از این آنزیم تضمین کننده حذف همه آلودگی های DNA نمی باشد و ممکن است DNAهای باقی مانده سبب جواب مثبت کاذب در نمونه ها شود.
مشکل چهارم آنزیم DNase قیمت بالای این آنزیم می باشد.
با توجه به مشکلات فوق در استفاده از آنزیم DNase بهتر است پرایمرها به صورتی طراحی شوند که در صورت وجود آلودگی DNA، پرایمرها الگوی DNA را تکثیر نکنند و به صورت اختصاصی الگوهای cDNA را تکثیر کنند.
آموزش نکات طراحی پرایمرهای intron inclusion ( intron-spanning) در ریل تایم با روش سایبر گرین ( Real Time PCR SYBR Green)
سه روش برای طراحی پرایمرها در بررسی بیان ژن ها وجود دارد. در روش اول (شکل 3) پرایمر فوروارد برروی یک اگزون و پرایمر معکوس برروی اگزون مجاور طراحی می شود. به شکل زیر توجه نمایید.
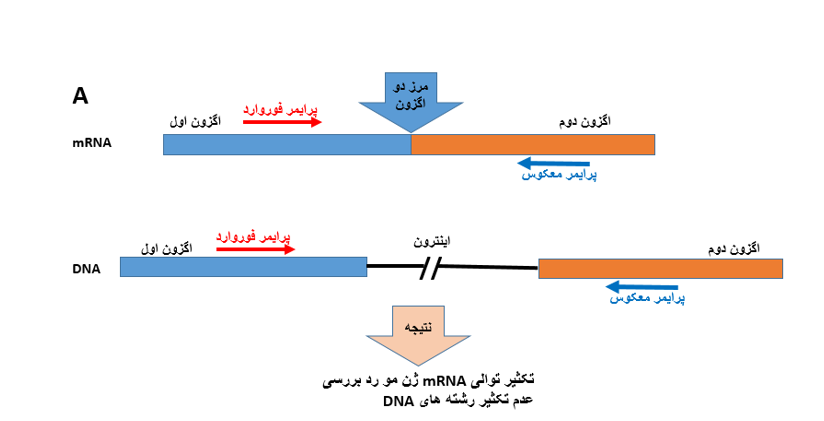
شکل 3: در روش فوق پرایمر فوروارد برروی یک اگزون و پرایمر معکوس برروی اگزون مجاور طراحی شده است. به دلیل اینکه در توالی DNA بین دو اگزون توالی اینترون وجود دارد، فاصله بین دو پرایمر در الگوی DNA به قدری زیاد می شود که این پرایمرها قادر به تکثیر الگوهای DNA نمی باشند. فرض نمایید در شکل فوق طول اینترون برابر با 3 هزار جفت باز باشد، حال اگر زمان extension واکنش ریل تایم را برروی 30 ثانیه تنظیم شده باشد، دیگر توالی 3هزار جفت بازی تکثیر نخواهد شد.
اما در این روش در الگوهای cDNA بین دو اگزون توالی اینترونی وجود ندارد بنابراین پرایمرها می توانند الگوی cDNA را تکثیر نمایند.
نکته: روش intron inclusion ( intron-spanning) بهترین و مطمئن ترین و آسانترین روش طراحی پرایمر برای بررسی بیان ژن ها می باشد اما در این روش باید بین دو اگزون انتخاب شده یک اینترون با طول حداقل 300 جفت باز وجود داشته باشد و هنگامی که طول اینترون های یک ژن از 300 جفت باز کمتر باشد استفاده از این روش طراحی پرایمر توصیه نمی گردد.
آموزش نحوه طراحی پرایمرهای exon-exon junction برای بررسی بیان ژن ها در ریل تایم با روش سایبر گرین ( Real Time PCR SYBR Green)
هنگامی که در یک ژن طول اینترون ها کوتاه باشد می توان از روش دیگری برای اختصاصی نمودن پرایمرها برای الگوهای cDNA استفاده نمود. به شکل 4 توجه نمایید.
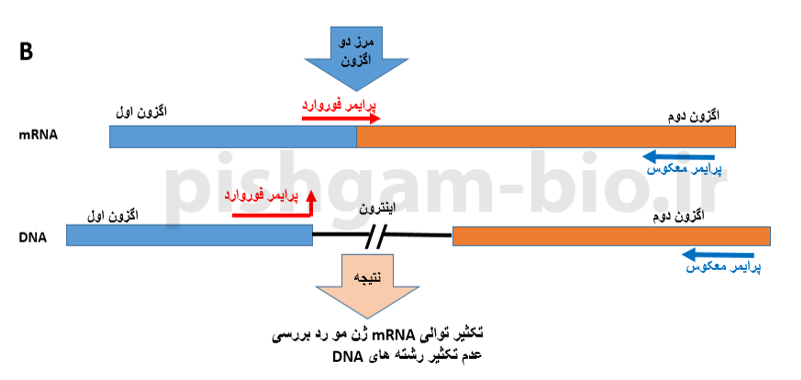
شکل 4: در طراحی پرایمر در روش جدید یکی از پرایمرها دقیقا برروی محل اتصال اگزون ها طراحی می گردد (در شکل فوق پرایمر فوروارد). در طراحی پرایمر به این روش باید توجه نمود که توالی سمت 3 پریم پرایمر که برروی اگزون دوم قرار می گیرد باید بین 3 تا 5 جفت باز طول داشته باشد(شکل 5)
و قرار دادن بیش از 6 جفت باز در سمت 3 پریم پرایمر اصلا توصیه نمی شود. به دلیل این که در این روش پرایمرها برروی مرز دو اگزون طراحی می گردند، به این روش exon-exon junction می گویند.
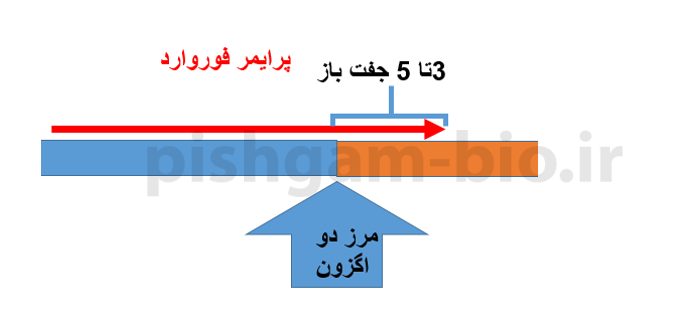
شکل 5: نحوه طراحی پرایمر در مرز دو اگزون برای بررسی بیان ژن ها
نکته: در روش exon-exon junction نیز پرایمرها پرایمرها قادر به اتصال به توالی DNA نمی باشند (به دلیل وجود اینترون ها) بنابراین آلودگی های DNA تکثیر نمی شوند.
نکته: روش exon-exon junction دارای یک مشکل بزرگ می باشد: در این روش تعداد مرزهای اتصال اگزون ها به یکدیگر کم می باشد بنابراین محل های انتخاب برای طراحی پرایمر محدود است. و ممکن است که هیچ یک از جایگاه ها دارای شرایط لازم بعدی برای طراحی پرایمر نباشند ( به عنوان مثال ممکن است پرایمر انتخاب شده دارای ساختار ثانویه نامناسب باشد).
آموزش نحوه طراحی پرایمرهای داخل اگزونی برای بررسی بیان ژن ها در ریل تایم با روش سایبر گرین ( Real Time PCR SYBR Green)
شکل 6 روش سوم در طراحی پرایمر برای بررسی بیان ژن ها را نشان می دهد.
شکل6: در این روش هر دو پرایمر فوروارد و معکوس برروی یک اگزون قرار دارد. گرچه در شرایط عادی طراحی پرایمر به این روش توصیه نمی گردد اما در برخی موارد ممکن است یک ژن فاقد اینترون باشد و یا طراحی پرایمر به دو روش قبلی ممکن نباشد. در این شرایط هر دو پرایمر برروی یک اگزون طراحی می گردند. باید توجه داشت در مواردی که پرایمرها به این روش طراحی می گردند باید با تیمار نمودن RNAهای استخراج شده با آنزیم DNase آلودگی DNA را تا حد امکان کاهش داد.
اصل دوم در طراحی پرایمر برای بررسی بیان ژن ها این است که بهتر است پرایمرها برروی اگزون های سمت 3 پریم mRNA طراحی شوند. دلیل این است که در سنتز cDNA آنزیم RT سنتز را از سمت 3 پریم mRNA آغاز می نماید. بنابراین سنتز cDNA سمت 3 پریم ژن ها با کیفیت بیشتری نسبت به سمت 5 پریم انجام می گیرد.
اصل سوم در طراحی پرایمر برای بررسی بیان ژن ها این است که طول قطعه تکثیر شونده (محصول ریل تایم) باید بین 90 تا 200 جفت باز باشد. البته در مقالات چاپ شده طول برخی محصولات PCR از این دامنه پیروی نکرده است اما توصیه می گردد طول قطعه در همین محدود باشد. طول کمتر سبب می گردد که قطعه تکثیر شده با دایمر پرایمر اشتباه گرفته شود. و طول بیشتر می تواند سبب افزایش تکثیر غیر اختصاصی و همچنین دو قله ای شدن منحنی ذوب محصول ریل تایم شود.
سه اصل ذکر شده در فوق نکات اختصاصی طراحی پرایمر برای بررسی بیان ژن ها می باشد. اما پرایمرهای طراحی شده باید دارای یک سری اصول عمومی نیز باشند . این اصول شامل ویژگی هایی مانند طول پرایمر، دمای Tm ، مقدار و درصد بازهای GC ، نحوه اتصال نوکلئوتیدهای انتهایی پرایمر می باشد.
در طراحی پرایمر این اصول زیر را باید همیشه مد نظر قرار دهیم: طول متوسط هر پرایمر بین 25-۱۸ جفت باز باشد، پرایمر با طول کوچک اتصال غیر اختصاصی را افزایش داده و پرایمر بزرگ سرعت هیبریداسیون را کم میکند.
میزان GC پرایمر های فوروارد و معکوس شبیه هم باشد که معمولا حدود ۶۰-40 درصد می باشد. پرایمر های مستقیم و معکوس نباید مکمل همدیگر باشند و با یکدیگر ساختار ثانویه تشگیل دهند، زیرا باعث تولید پرایمر دایمر می شود که به ویژه در Real time PCR مشکل ایجاد می کند.
قرار دادن حداقل یک باز G یا C در انتهای ۳ پریم پرایمر سبب افزایش کارایی پرایمر می گردد اما قرار دادن یک باز A یا T سبب افزایش اختصاصیت پرایمر می گردد.
باید دقت کرد که انتهای ۳ پریم غنی از C و یا G (و یا هر دو) نباشد که سبب کاهش اختصاصیت و اتصال های اشتباه به ژنوم می شود.
باید تا حد امکان از توالی های پالیندرومیک (برای مثال GTTAAC ) در پرایمر جلوگیری کرد، زیرا احتمال تشکیل پرایمر دایمر را افزایش می دهند.
در پرایمرهای طراحی شده در حد امکان تعداد پورین ها با پریمیدین ها برابر باشد.
از توالی های تکراری (برای مثال AAAAAA ) در پرایمر، به ویژه در ۳ پریم جلوگیری شود و دمای Tm دو پرایمر نزدیک هم باشد.
بنابراین اصول عمومی طراحی پرایمر عبارتند از:
- طول پرايمر ها بايد بين 18 تا 25 نوکلئوتيد باشد.
- دماي Tm دو پرايمر فوروارد و معکوس بايد نزديک به هم باشد.
- پرايمر ها بايد از نظر درصد GC در حد نرمال باشند(حدود 40تا60 درصد).
- پرايمر ها نبايد ساختار سنجاق سری تشکيل دهند.
- پرايمر ها تشکيل Self-Dimer و یا Hetero-Dimer ندهد.
در بخش های فوق، مقدمات طراحی پرایمر را برای بررسی ژن ها در ریل تایم به روش سایبرگرین شرح دادیم. فایل آموزش پیشرفته طراحی پرایمر برای متیلاسیون شامل آموزش کامل طراحی پرایمرهای exon-exon junction و intron-spanning برای ریل تایم به روش سایبرگرین (real time pcr) با استفاده از نرم افزارهای primer3 و primer blast می باشد که می توانید لیست کامل آموزش های فایل پیشرفته را در فهرست ارائه شده در ابتدای همین آموزش مشاهده نمایید.
برای تهیه آموزش پیشرفته طراحی پرایمر برای ریل تایم می توانید از لینک زیر اقدام نمایید.
https://pishgam-bio.ir/%d9%81%d8%b1%d9%88%d8%b4%da%af%d8%a7%d9%87/












دیدگاهتان را بنویسید