با پیشرفتهای ایجاد شده در زمینههای نانو تکنولوژی و بیوانفورماتیک روشهای جایگزینی جهت افزایش سرعت و افزایش بازدهی تعیین توالی DNA ابداع شده است. اصطلاح Next Generation sequencing جهت توصیف مجموعهای از فن آوریها با قدرت بالاتر نسبت به توالییابی به شیوه سنگر مورد استفاده قرار می گیرد که شامل نسل دوم از دستگاههای تعیین توالی بوده که قطعات را بصورت همزمان و با سرعت بالا تعیین توالی می کنند. برتری این روش ها نسبت به روش تعیین توالی بر پایه ختم زنجیر این است که این تکنیکها بدون نیاز به ژل و در حین سنتز DNA، توالی DNA را ثبت میکنند. از طرفی این سیستم نیازی به کلونینگ نداشته و از پروسه PCR جهت از دیاد قطعه مورد نظر استفاده می کند. این امر منجر به افزایش سرعت و بازدهی تعیین توالی DNAمی گردد. این تکنیکها شامل:
توالی یابی پایرو ( تکنیک پایرو سکوئنسینگ Pyrosequencing)
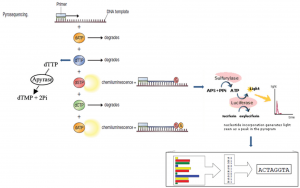
تکنیک پایرو سکوئنسینگ توسط دکتر مصطفی رونقی و همکارانش ابداع گردید. این روش نیز نیازمند DNA تک رشته بعنوان الگو می باشد که از طریق دناتوراسیون محصولات PCR بدست می آید. در این تکنیک پس از اتصال پر ایمر به توالی مکمل، سنتز رشته جدید بواسطه آنزیم پلیمراز آغاز می گردد. در واکنش های پلیمریزاسیون به ازای هر پیوند فسفودی استر یک گروه پیر و فسفات (ppi) و یک یون هیدروژن آزاد می شود. آنزیم ATP سولفوریلاز از این گروه پیروفسفات به همراه آدنوزین فسفو سولفات (APS) موجود در مخلوط واکنش به منظور تولید ATP استفاده می کند. ATP تولیدی نیز انرژی مورد نیاز برای آنزیمی موسوم به الوسيفراز را مهیا میکند. این آنزیم، لوسیفیرین را اکسید کرده و منجر به تولید اکسي لوسیفیرین میشود که در نتیجه این واکنش نور تولید می گردد. این سیگنال نوری توسط دوربینهای حساس به سیگنالهای ساطع شده موسوم به CCD بصورت یک پیک ثبت میشود و از آنجاکه نوکلئوتیدهایی که در هر مرحله اضافه می شوند مشخص و یکسان هستند، می توان بر اساس تولید این پیک، توالی DNA را در حین سنتز بدست آورد. از طرفی در هر مرحله جهت اضافه کردن نوکلئوتیدهای جدید میبایست نوکلئوتیدهای قبلی از محیط حذف شوند که این کار توسط آنزیمی موسوم به آپیراز انجام می پذیرد. آپیر از یک آنزیم نوکلئوتیداز می باشد. در شکل ۴ نمایی از این پروسه نشان داده شده است. فرم تجاری روش پایر وسکو تنسینگ در سال ۲۰۰۵ توسط شرکت Life Science ۴۵۴ ارائه شد که از آن تحت عنوان تعیین توالی به روش ۴۵۴ Pyrosequencing نیز یاد میشود. در این تکنیک، ابتدا DNA ژنومی به قطعات ۳۰۰ إلى ۵۰۰ جفت بازی شکسته شده و سپس بصورت تک رشته به مهره های فلزی متصل می گردد. بدین صورت که در ابتدا قبل از دناتوراسیون، به دو سمت این قطعات آداپتور متصل می شود. سپس این قطعات تک رشته ای می شوند. آداپتور دو نقش مهم را ایفا می کند. اولا در اتصال قطعات تک رشته به مهره های فلزی کوچک نقش داشته چرا که یکی از این آداپتورها در انتهای ۵ خود حاوی یک گروه بیوتین میباشد و از آنجاکه این دانه های فلزی با استرپتو آویدین پوشیده شده اند، اتصال این قطعات تک رشته به دانه ها بواسطه تمایل بالای اتصال استرپتو آویدین به بیوتین انجام می گیرد. این پروسه به گونهای طراحی شده است که به هر مهره فلزی یک قطعه DNA تک رشته متصل می شود. در نهایت هر کدام از این مهرهها، در درون یک قطره آب در امولسیون روغن قرار می گیرند که در اصطلاح، این فضا میکرور آکتور نامیده می شود و واکنش PCR بمنظور تکثير قطعه DNA درون این میکرور آکتور صورت می گیرد. میکرو رآکتورها تمامی عوامل مورد نیاز جهت تکثیر قطعه DNA تک رشته را دارا می باشند و بصورت فیزیکی بواسطه سد ایجاد شده توسط جزء روغن امولسیون از یکدیگر جدا می گردند. به این نوع تکثير ، PCR- امولسیون نیز گفته می شود. در این مرحله آداپتورها دومین نقش خود را ایفا می نمایند بطوریکه آداپتورها بعنوان جایگاهی برای اتصال پرایمرها و در نتیجه تکثير قطعه مورد نظر، عمل می کنند. از آنجاکه این تکنیک نیازمند DNA الگو تک رشته میباشد، پس از اتمام مرحله تکثير ، محصولات PCR باید دناتوره شوند. بدین ترتیب پس از تکثیر بر روی هر دانه در حدود ۱ میلیون قطعه DNA تک رشته جهت تعیین توالی ایجاد می گردد. هدف از تکثیر، افزایش قطعه مورد نظر جهت تقویت شدت سیگنال و بالطبع شناسایی بهتر توسط detector در مرحله تعیین توالی می باشد. در مرحله بعد امولسیون را شکسته و محتویات درون آن را به داخل چاهکهای پیکولیتری موجود در سطح یک پلیت منتقل می کنند. شرایط طوری انتخاب می گردد که در داخل هر چاهک تنها یک دانه می تواند قرار گیرد. سپس آنزیمهای سولفوریلاز، لوسیفر از و آپیر از به همراه سایر عوامل مورد نیاز برای تکثیر به مخلوط واکنش افزوده می شود و بدین ترتیب پایر و سکوئنسینگ در هر چاهک بطور مستقل به شیوهای که در بالا توضیح داده شد انجام می گیرد. این تکنیک قادر به توالییابی بیش از ۱ میلیون نمونه بطور همزمان می باشد.
شکل 4- نمایی از تکنیک پایروسیکوئنسینگ
توالی یابی نسل آینده : آموزش تکنیک (Semiconductor Sequencing (lon Torrent برای توالی یابی ژنوم ، DNA و RNA (cDNA)
این تکنیک در سال ۲۰۱۰ توسط شرکت Life Technologies ارائه شد. این پروسه به تکنیک پایروسکوئنسینگ بسیار شبیه می باشد با این تفاوت که در این تکنیک از تغییرات pH که بواسطه رها شدن یون ۲۰ به هنگام سنتز رشته جدید در محیط ایجاد می گردد، برای تشخیص نوکلئوتیدها استفاده می شود. مراحل آماده سازی این تکنیک، مشابه با روش پایروسکوئنسینگ می باشد (قطعات در این روش به طول ۲۰۰ جفت باز می باشند با این تفاوت که در نهایت مهره های فلزی که حاوی بیش از یک میلیون DNA تک رشته می باشند را به داخل میکرو ولهایی انتقال می دهند که در زیر این چاهکها یک حسگر فوق حساس یونی تعبیه شده است که سریع تغییرات pH را ثبت میکند و در نهایت این تغییرات بصورت یک پیک در نمایشگر ظاهر میشود. از آنجاکه در این روش نیز در هر مرحله نوکلئوتیدهای مشابه و مشخص به مخلوط واکنش افزوده میشود، می توان بر اساس تولید این پیک، توالی DNA را در حین سنتز بدست آورد. در شکل ۵ مراحل این پروسه نشان داده شده است.
شکل 5- تعیین توالی به روش Ion Torrent
توالی یابی نسل جدید : تکنیک (illumina (solexa برای توالی یابی ژنوم ، DNA و RNA (cDNA)
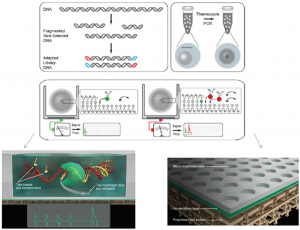
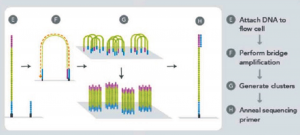
این روش توسط شرکت ILumina در سال ۲۰۰۷ ارائه گردید. در ابتدا ژنوم مورد نظر به قطعاتی از DNA تا طول ۱۰۰ الی ۱۵۰ جفت باز شکسته میشود و قبل از تک رشته ای شدن به دو سمت این قطعات آداپتور متصل میگردد. سپس این قطعات تک رشته ای شده و بصورت رندوم بر روی یک سطح جامد توزیع میگردند. این سطح جامد بطور متراکم با توالی های مکمل آداپتور پوشیده شده است در نتیجه هر قطعه از DNA تک رشته از یک انتهای خود بواسطه اتصال به توالیهای مکمل آداپتور بر روی این سطح جامد ثابت می گردد. در مرحله بعد انتهای آزاد این قطعات نیز به توالی های مکمل آداپتور متصل شده و در نتیجه منجر به تشکیل یک پل میشوند. سپس تکثیر این پل بواسطه افزودن عوامل مورد نیاز برای PCR آغاز می گردد که در اصطلاح به این پروسه، bridge amplification or Bridge PCR گفته می شود. در این پروسه، توالیهای مکمل آداپتور بعنوان پر ایمر عمل می کنند. پس از اتمام هر سیکل، DNA دو رشته ای حاصل دناتوره شده تا دوباره پروسه PCR انجام گیرد. این پروسه تا زمانیکه مجموعههایی متراکم از قطعه مورد نظر ایجاد شود، تکرار می گردد چراکه هدف در این مرحله افزایش تعداد قطعه مورد نظر به جهت دستیابی به شدت سیگنال بیشتر میباشد. از آنجا که این تکنیک نیز نیازمند DNA تک رشته بعنوان الگو می باشد، پس از اتمام مرحله تکثير، محصولات PCR باید دناتوره شوند. در این حالت مجموعههایی در حدود ۱۰۰۰ کپی از DNA تک رشته بصورت رندوم بر روی سطح جامد ایجاد می گردد که در اصطلاح به هر کدام از این مجموعهها، DNA Polony گفته می شود که هر یک از این مجموعهها نیز متعلق به یک قطعه DNA میباشند. در این روش می توان بطور همزمان حداقل ۴۰ میلیون پولونی که هرمجموعه متعلق به یک قطعه DNA ست را تعیین توالی کرد. مراحل کار در شکل ۶ نشان داده شده است. در نهایت جهت تعیین توالی، عوامل مورد نیاز برای پروسه PCR که شامل پرایمر، نوکلئوتیدهای ختم دهنده و DNA پلیمراز می باشد را به سطح این صفحات جامد می افزایند. پر ایمرهاییکه در این مرحله مورد استفاده قرار می گیرد، مکمل توالیهای آداپتور بوده که به انتهای آزاد قطعات تک رشته در هر کلاستر متصل شده و بواسطه عملکرد آنزیم پلیمر از منجر به سنتز رشته جدید میشوند. در این تکنیک از نوع خاصی از نوکلئوتیدهای ختم دهنده موسوم به 3-blocked reversible terminatornucleotides- استفاده می شود که هر کدام از این نوکلئوتیدها، با یکی از ۴ رنگ فلورسانس (سبز، قرمز، آبی و نارنجی) لیبل می شوند. برخلاف ddNTPهای مورد استفاده در تکنیک سنگر، این نوکلئوتیدها بصورت برگشت پذیر عمل کرده و پس از شناسایی شدن لیبل رنگی آنها بواسطه detector، پلی مریزاسیون رشته در حال سنتز متوقف نمیشود و ادامه می یابد چراکه پس از آزاد شدن لیبل رنگی و شناسایی آن بواسطه دستگاه، گروه terminator که انتهای ۳ این نوکلئوتیدها را بلوکه کرده نیز جدا میشود و در نتیجه اثر مهاری آن برداشته شده و گروه OH-۳ نمایان می گردد. این امر منجر به ادامه تکثیر رشته در حال سنتز میشود. در حقیقت نقش این نوکلئوتیدهای ختم دهنده ایجاد یک وقفه کوتاه در تکثیر رشته در حال سنتز جهت شناسایی لیبل رنگی آن نوکلئوتید و در نتیجه مشخص شدن نوع آن نوکلئوتید جهت تعیین توالی می باشد. در این تکنیک نوکلئوتیدها بر خلاف روش پایروسکوئنسینگ و یا lon Torrent که بصورت مجزا از یکدیگر در هر مرحله افزوده می شوند، بصورت همزمان به مخلوط واکنش در هر مرحله اضافه می گردد که بر اساس ليبلهای فلورسانس متفاوتشان از یکدیگر متمایز میشوند. تفاوت دیگر این است که در روش Solexa، پس از اتمام هر مرحله یک شستشو انجام می گیرد تا نوکلئوتیدهایی که در زنجیره در حال ساخت وارد نشدهاند از محیط حذف گردند و سپس مرحله بعدی آغاز می شود در صورتیکه در تکنیک پایروسکوئنسینگ و یا lonTorrent از آنزیم آپیر از بدین منظور استفاده می گردد.
شکل 6- نمایی از Bridge PCR
تکنیک ABI (Applied Biosystems) solid (sequencing by Oligo Ligation and Detection) system برای توالی یابی ژنوم ، DNA و RNA (cDNA)
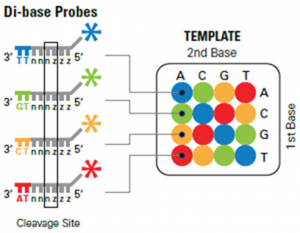
این تکنیک در سال ۲۰۰۷ توسط شرکت Applied Biosystems ارائه شد. اساس این روش بر پایه اتصال پروبهای الیگونوکلئوتیدی لیبل شده با ۴ رنگ مختلف فلورسانس (قرمز، آبی، نارنجی و سبز) میباشد که به موجب آن، هر پروب موقعیت دو باز را در زنجیره مشخص می کند. در این تکنیک قطعاتی از DNA در حدود ۳۵ الی ۷۵ جفت باز ایجاد شده و به دو انتهای این قطعات آداپتورها متصل می شود. سپس این قطعات تک رشته شده و بواسطه تمایل اتصال بیوتین به استرپتو آویدین به سطح دانههای فلزی متصل می گردند. در مرحله بعد هر کدام از این دانهها که حاوی یک قطعه DNA تک رشته میباشند، در درون یک قطره آب در امولسیون روغن قرار می گیرند و واکنش PCR- امولسیون جهت تکثیر قطعه DNA درون این میکرو رآکتور انجام میشود. در نهایت پس از مرحله تکثیر و دناتوراسیون، هر کدام از دانههای فلزی که در حدود ۱ میلیون قطعه DNA تک رشته به آنها متصل میباشد، جهت تعیین توالی به یک اسلاید شیشهای انتقال داده میشوند. سپس پرایمرها که مکمل توالی آداپتور بوده و مخلوطی از پروبهای الیگونوکلئوتیدی به این سطح افزوده میشود. در مرحله اول پر ایمر به آداپتور متصل می گردد. سپس مخلوطی از الیگونوکلئوتیدهای اکتامر که انتهای آنها با رنگهای فلورسانس مختلف لیبل شده است به قسمتهای مکمل خود بر روی رشته الگو متصل می شوند. اتصال این قطعات به یکدیگر بواسطه آنزیم لیگاز صورت می گیرد. بطبع این الیگو نوکلئوتیدها باید توالی متنوعی داشته باشند تا امکان حضور توالی مکمل بر روی رشته DNA افزایش یابد. برای این امر از di-base probeها استفاده می شود. همانطور که از اسم این پروبها مشخص است، این پروب ها در ابتدای خود حاوی دو باز مشخص میباشند و مابقی بازها از توالیهای دجنره و یونیورسال می باشند که با حرف 1 و 2 نشان داده میشوند. مثالی از این پروب ها در شکل ۷ نشان داده شده است.
شکل 7- نمایی از di-base probe
ویژگی این پروب ها مربوط به ۲ باز اول میباشد و حتی لیبل رنگی که به انتهای این پروب ها متصل می گردد بسته به این دو باز تعیین میشود. بطوریکه یک الفبای دو حرفی و ۱۶ حالته بر اساس این دو باز در روش Solid موجود میباشد که از هر دو حرف نوکلئوتیدی یک کد یا رنگ خاصی حاصل میشود. الفبای فوق ۴ قانون دارد که شامل موارد زیر میباشد:
۱) قانون معكوس: دو نوکلئوتید مختلف در هر دو حالت ترکیبی خود، یک رنگ یا کد مشترک دارند. مثلا دو نوکلئوتید آو c در هر دو حالت CT و معکوس آن TC، به رنگ نارنجی و کد ۲ شناخته می شوند.
۲) قانون مکمل: نوکلئوتیدهای ۲ بازی مکمل نیز رنگ و کد یکسان دارند. مثلا در باز CA و مکمل آن CT هر دو دارای رنگ سبز یا کدامی باشند.
٣) قانون مکمل معكوس: با توجه به دو قانون قبل، نوکلئوتیدهای ۲ بازی در دو حالت مکمل و معکوس نیز رنگ و کد یکسان دارند. مثلا توالی آA و حالت مکمل معکوس آن یعنی CG هر دو دارای رنگ قرمز یا کد ۳ هستند.
۴) دو نوکلئوتید یکسان مثلا AA، دارای کد 0 و رنگ آبی می باشند. در جدول ۱ این تقسیم بندی آمده است.
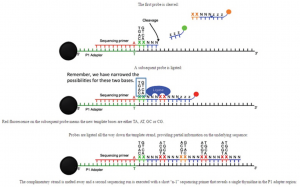
تعیین توالی بر پایه Ligation در ۵ سیکل که هر سیکل نیز شامل چندین مرحله میباشد، بواسطه پر ایمرهای مختلف انجام می گیرد. بدین ترتیب که در هر سیکل یک نوکلئوتید از پرایمر مربوطه کم میشود یعنی در دور اول از پرایمر n، در دور دوم از پرایمر ۱-n در دور سوم از پرایمر ۲-n، در دور چهارم از پرایمر 3-n و در دور پنجم از پرایمر ۴-n استفاده می گردد. در سیکل اول پس از اتصال پرایمر n به توالی آداپتور، یک مجموعه چهارتایی از این پروب ها به مخلوط واکنش اضافه می شود که برای اتصال به قطعه پرایمر با یکدیگر رقابت می کنند. در نهایت پس از اتصال یکی از این پروبها، لیبل فلورسانس مربوط به این پروب به همراه ۳ باز آخر بصورت آنزيماتیک جدا میگردد (همانطور که در شکل ۹ نشان داده شده است این پروب ها حاوی یک cleavage site می باشند که پس از اتصال به توالی مکمل، پروب از این ناحیه بصورت آنزیماتیک شکسته میشود). بواسطه جدا شدن لیبل رنگی، نوری ساطع می شود که توسط دستگاه ثبت میگردد. این مرحله آنقدر تکرار میشود تا رشته جدید بطور کامل سنتز شود. نکته قابل توجه این است که قبل از ورود به هر مرحله، پروب های متصل نشده بواسطه شستشو از محیط حذف می گردند تا اختلالی در مراحل بعدی ایجاد نکنند. پس از اتمام سیکل اول وارد سیکل دوم می شویم. قبل از شروع سیکل دوم، رشته حاوی پرایمر n بواسطه حرارت از رشته الگو جدا شده و از طریق شستشو از محیط حذف می گردد و بدین ترتیب واکنش با پرایمر ۱-۳ در سیکل دوم آغاز میشود. این واکنش نیز همانند سیکل اول تا تکمیل شدن رشته در حال سنتز بواسطه پروب های مختلفی که بصورت مرحلهای (در هر مرحله یک مجموعه ۴ تایی) به واکنش افزوده می شود، ادامه مییابد. نمایی از این تکنیک در شکل ۸ نشان داده شده است.
شکل Sequencing by Ligation -8
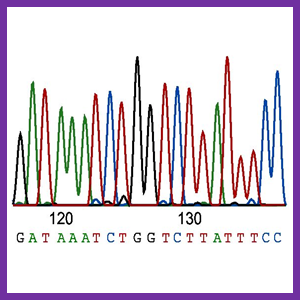
در نهایت با تلفیق تمام دادهها در ۵ سیکل می توان به توالی رشته مورد نظر دست یافت که در شکل ۱۱ و ۱۲ این وقایع نشان داده شده است. این تکنیک قادر به توالییابی بیش از ۵۰ میلیون نمونه بطور همزمان می باشد .

















یک پاسخ
سلام وقت بخیر منبع این اطلاعات رو میتونم داشته باشم ؟