آشنایی جامع با مکانیسم متیلاسیون dna ،جایگاه و عملکرد methylation در فعالیت ها و فرایندهای زیستی و ژنتیکی
روشهای بررسی متیلاسیون ژنها و DNA که مبتنی بر طراحی پرایمر و PCR می باشند در این قسمت آموزش داده خواهند شد.
تعریف متیلاسیون (DNA Methylation) DNA : متیلاسیون یعنی چه ؟
از دهه قبل مشخصی شده که DNA متعلق به موجودات گوناگون، علاوه بر 4 باز استاندارد، حاوی بازهای متیله N- متیل آدنین، C- متیل سیتوزین و N- متیل سیتوزین است (شکل 1). باید به این نکته اشاره نمود که این بازهای متیله شده، اجزاء تشکیل دهنده طبیعی DNA بوده و با تغییرات شیمیائی بازها نظیر آلکیلاسیون و آسیبهای اکسیداتیو DNA متفاوت هستند. متیلاسیون DNA در ویژگیهای واتسون – کریک بازهای آدنین و سیتوزین اختلالی ایجاد نمیکند، بهطوریکه گروه متیل در شیار بزرگ DNA قرارگرفته و بهراحتی بهوسیله پروتئینهای برهمکنشی کننده با DNA قابل شناسایی است.
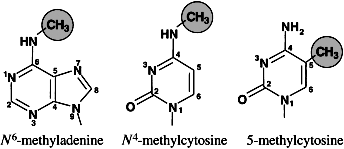
شکل 1- نمایشی ساختار شیمیایی بازهای متیله شونده در DNA
بدین وسیله متیلاسیون، اطلاعات اضافی به DNA القاء مینماید، بهگونهای که کدگذاری نشده و میتوان بازهای متیله را بهعنوان متیلاسیون DNA ارتباط نزدیکی باهمانندسازی DNA دارد و به استثنای رشته جدید، این دو فرآیند بهصورت همزمان انجام میپذیرند. در پروکاریوت ها، متیلاسیون DNA در هر دواحد سیتوزین و آدنین رخ میدهد، همچنین در فرآیندهایی نظیر تنظیم بیان ژن، ترمیم DNA، کنترل تکثیر سلول و دفاع علیه DNA خارجی نقشی دارد و این در حالی است که در یوکاریوت ها، متیلاسیون بهصورت هماهنگ با سایر تغییرات اپی ژنتیک، در تنظیم بیان ژن و ساختار کروماتین نقش ایفاء مینماید. در پستانداران، متیلاسیون DNA بر روی کربن ۵ سیتوزین قرارگرفته در دی نوکلئوتید CpG و بندرت در توالیهای نامتقارن (CpNpG،CpT، CpA، CC(a/t)GG) انجام میشود، حال متیلاسیون سیتوزین در همه یوکاریوت ها انجام نمیشود، بهگونهای که در ساکارومیسی سروزیه و بسیاری از بیمهرگان نظیر نماتدها متیلاسیون DNA مشاهده نمیگردد. در حشرات مانند مگس زنبور عسلی، سطح پائینی از متیلاسیون DNA وجود دارد.
در ژنوم هاپلوئید انسان تقریباً ۲۸۲۳۳۹ دی نوکلئوتید CpG وجود دارد که ۷۰ درصد (بسته به گزارشیهای مختلف، بین ۱۰ تا ۹۰ درصد) آنها در سلولهای نرمال متیله میشوند. فقط ۷ درصد از کل CpGها، در جزایر CpG قرارگرفتهاند که اغلب آنها در سلولهای نرمال غیرمتیله هستند. از استثنائات در این مورد میتوان به متیلاسیون نرمال دی نوکلئوتیدهای متعلق به تعداد کمی از جزایر CpG موجود در کروموزوم X غیرفعال (خاموش شدن تصادفی یکی از کروموزوم های X در هر یک از سلولهای سوماتیک نرمال در پستانداران ماده)، ژنهای حک گذاری شده (بیان یا عدم بیان ژنهای معین منطبق با منشأ والدینی) و اختصاصی بافت و علاوه براین، DNA پری سانتریک توالیهای ماهوارهای سانترومر کروموزوم های ۱ و ۱۱، نواحی بین ژنی و توالیهای تکراری اشاره نمود. در واقع ۵ درصد از CpGهای موجود در ژنوم، در نواحی تکراری DNA قرار دارند و این نسبت بزرگی از کل ۵- متیل سیتوزین ژنوم را به خود اختصاص میدهد. باز تغییریافته ۵- متیل سیتوزین، به دلیل ناپایداری گروه آمین موقعیت 6، فوقالعاده جهش پذیر بوده و میتواند متحمل دآمیناسیون خود به خودی و جایگزینی با تیمین شود. از آنجا که این موتاسیون توسط سیستم ترمیم DNA قابل شناسایی نیست، به تجمع موتاسیون از نوع C-T منجر میگردد. این مشاهده که میتواند علت کاهش ۵ برابری دی نوکلئوتید CpG (1 به ۸۰) را در مقایسه با نسبت مورد انتظار (۱ به 16) توجیه نماید، سرکوب CpG-SuppreSSlOn) CpG) نامیده میشود.
تعریف جزایر CpG Islands) CpG ) در ژنوم موجودات
علیرغم تنوع موجود در توالی پروموترها، ژنهای رونویسی شونده توسط RNA پلیمراز II را مطابق با توزیع دی نوکلئوتید CG ( 5-CpG-3)، میتوان به دو گروه تقسیم بندی نمود. فراوانی CpG در کلاس اول همسان با میانگین ژنوم است (۱ از هر ۱۰۰ دی نوکلئوتید). این کلاس غالباً شامل ژنهایی است که بیان آنها به تعداد معینی از انواع سلولها محدود میشود. در مقابل، انتهای ۵ ژنهای متعلق به کلاس دوم توسط ناحیهای تقریباً یک کیلو بازی (۵۰۰ تا ۵۰۰۰ جفت باز) احاطهشده که فراوانی CpG در آنها بیش از ۱۰ برابر میانگین ژنوم است (۱ از هر ۱۰ دی نوکلئوتید) و علاوه بر این، محتوی CpG آنها ۱۰ تا ۷۰ درصد و نسبت CpG/GpC در آنها حداقل 6/۰ است. این نواحی شدیداً اختصاصی، جزایر CpG نامیده میشوند. درصد C+G در جزایر CpG ژنوم انسان و موش، به ترتیب تقریباً 6۷ و 64 درصد میباشد. با این حال قابل ذکر است که دی نوکلئوتید CpG در جزایر CpG مذکور، عليرغم فراوانی شان غیرمتیله باقی میمانند. ۷۰ درصد جزایر CpG با ژنها همراه هستند و بیش از نیمی از آنها در انتهای ‘۵ ژنها قرار دارند و این دخالت احتمالی آنها در تنظیم رونویسی و پتانسیل کاربرد آنها را در مکانیابی ژنها در ژنوم پیشنهاد مینماید.
بندرت جزایر CpG غیرمعمول نیز در بدنه و حتی در نواحی ۳ پریم ژنها مشاهده میشوند که مستعد به متیلاسیون هستند. هم اکنون پس از ۲۰ سال از شناسایی، هنوز این نواحی اختصاصی، قابلاعتمادترین شاخصی برای ردیابی و شناسایی نواحی پروموتر در ژنوم پستانداران محسوب میشوند. تجزیه کروماتین در مقیاس کل ژنوم نشان میدهد که این نواحی، ویژگیهای معمول کروماتین فعال را دارند. این خصوصیات شامل متیلاسیون هیستونهای H3 و H4، فقدان هیستون H1 و ناحیه خالی از نوکلئوزوم است. نتایج به دست آمده از برخی مطالعات، از برآورد تقریبی تعداد ۵۰۰۰ و ۳۷۰۰۰ جزیره CpG به ترتیب در ژنوم انسان و موش حکایت دارد اگر چه تجزیههای رایانهای، این مقادیر را به ۲۹۰۰۰ و ۱۵۰۰۰ کاهش داده است. در حدود ۵۰ تا ۱۰ درصد ژنهای انسان، در نواحی تنظیمی خود واجد جزایر CpG هستند که این میزان، ژنهای خانهدار ( House Keeping genes) و در حدود نیمی از ژنهای اختصاصی بافت (Tissue-specific genes را شامل میگردد (شکل 2).
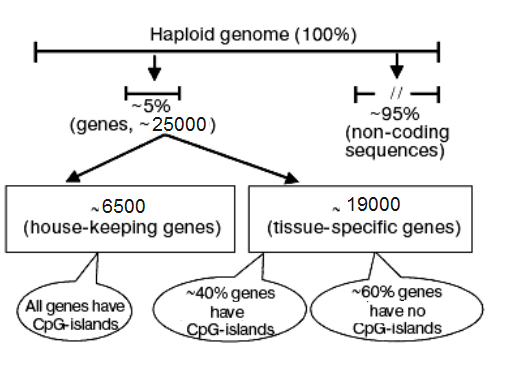
شکلی 2- نمایشی انتشار جزایر CpG در ژنوم انسان: ژنوم هاپلوئید انسان متشکل از ۱۰ ×۳/۲ باز و تقریباً حاوی 25۰۰۰ ژن است. قسمتی از آنها (تقریباً 65۰۰ ژن)، ژنهای خانهدار را تشکیل میدهند و مابقی عملکرد اختصاصی دارند. اکثر ژنهای متعلق به دسته اول و 40 درصد ژنهای دسته دوم، دارای جزایر CpG در نزدیکی توالی تنظیمیشان هستند. از این رو، 13۰۰۰ جزیره CpG بهصورت متعادل در بین ژنهای پایهای و اختصاصی پخش شدهاند.
دو نوع متیلاسیون وجود دارد: هیپومتیلاسیون و هیپرمتیلاسیون. هیپرمتیلاسیون DNA در عناصر پروموتور سبب جلوگیری از بیان ژن گردیده و برای دامنه گستردهای از فعالیتهای سلولی مانند پایداری و حفاظت ژنوم، ایمپرینتینگ، غیرفعالسازی کروموزوم X، تنظیم بیان ژنهای اختصاصی بافتها، سرطان و پیری ضروری است. هیپومتیلاسیون DNA نقش مهمی در ایجاد سرطان بازی میکند. الگوی متیلاسیون ژنوم در طی تکامل به علت فرآیندهای دینامیک شامل متیلاسیون de novo و دمتیلاسیون تغییر میکند. سلولهای تمایز یافته دارای الگوی ثابت و یکنواختی از متیلاسیون DNA است که نسخه برداری ژنهای ویژه بافتها را تنظیم میکنند. در این مبحث مهمترین تغییر اپی ژنتیک که بهصورت مستقیم سبب تغییر شیمیایی DNA میشود، یعنی متیلاسیون DNA موردبررسی قرار میگیرد.
نقش متیلاسیون DNA در سرکوب رونویسی ژنها
در ارتباط با دخالت متیلاسیون DNA در سرکوب رونویسی ژنها، دو مکانیسم پیشنهاد شده است: نخست اینکه، متیلاسیون DNA به طور مستقیم از اتصال فاکتورهای رونویسی (Transcription Factors) حساس به متیلاسیون مانند c- AP2 E2F cMyb SP1 ATF/CREB ETS NFkB Myc/Myn به نواحی اتصالیشان در پروموتر ژنها ممانعت به عمل میآورد. در این مکانیسم باید دی نوکلئوتیدهای CpG، درون جایگاههای اتصال این فاکتورهای رونویسی حساسی به متیلاسیون قرار داشته باشند. مکانیسم دیگر این است که اتصال فاکتورهای دارای دمین MBD به DNA متیله شده، سبب مهار رونویسی ژنها میشود (شکل 3).
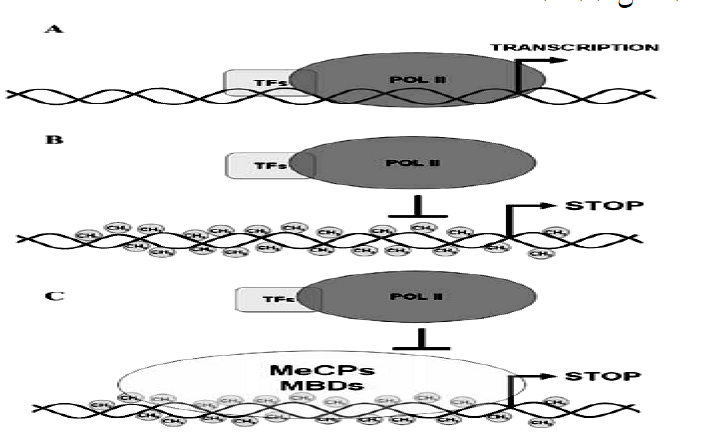
شکل 3– سرکوب رونویسی بهواسطه متيلاسيون دی نوکلثوتيدهای CpG. : (A RNA پلیمراز II و فاکتورهای رونویسی بهمنظور آغاز رونویسی، به توالی پروموتر متصل میشوند. (B) متیلاسیون CpGs درون جایگاههای اتصال قرارگرفته در پروموتر، سبب مهار مستقیم جذب فاکتورهای رونویسی میشوند. (C) اتصال فاکتورهای واجد دمین اتصال به MBD) m5-CpG و MeCPS)، از اتصال فاکتورهای رونویسی به توالی پروموتر ژنها ممانعت به عمل میآورند.
متیلاسیون DNA و فرآیند سرطانزایی
سرطان یک بیماری چند مسیری با ضایعات ژنتیکی است و تمام این ضایعات برای ایجاد یک تومور کاملاً تثبیتشده موردنیاز است. بهعلاوه وضعیت یکسانی نیز برای ضایعات اپی ژنتیک وجود دارد. باوجود گذشت ۲۵ سال از درک نقش متیلاسیون DNA در فرآیند خاموشی ژن، کماکان تحقیقات پیرامون این موضوع، موردتوجه بسیاری از محققین است. در مقایسه با سلولهای طبیعی، سلولهای توموری دو نوع اصلی از ضایعه را در الگوی متیلاسیون نشان میدهند: هیپومتیلاسیون DNA معمولاً توالیهای تکراری و هیپرمتیلاسیون DNA جزایر CpG را هدف قرار میدهند .
هیپومتیلاسیون DNA (DNA Hypomethylation)
سطح پائین متیلاسیون DNA در تومورها در مقایسه با بافتهای طبیعی، یکی از نخستین تغییرات اپی ژنتیک شناختهشده در سرطانهای انسانی است. فقدان متیلاسیون DNA عمدتاً به علت هیپومتیلاسیون توالیهای تکراری و دمتیلاسیون نواحی کدگذار و اینترون DNA است که رونویسی نابجا از ژنها را سبب میشود.
مطالعات متیلاسیون DNA با استفاده از روش Genomic Microarray در مقیاس کل ژنوم، در سلولهای توموری هیپومتیلاسیون وسیعی را در نواحی ضعیف ازنظر وجود ژنها نشان میدهد. در حین ایجاد سرطان هیپومتیلاسیون بهصورت یک ضایعه پیشرونده از تکثیر خوشخیم تا شکل متاستازی سرطان را نشان میدهد.
هیپرمتیلاسیون DNA و بروز سرطان.
علاوه بر هیپومتیلاسیون، هیپرمتیلاسیون در جزایر CpG ژنهای سرکوبکننده سرطان نیز میتواند یکی از دلایل اصلی بروز سرطانها باشد.
تجربیات نشان داده که هیپرمتیلاسیون جزایر CpG، مکانیسم غیرفعال شدن ژن مهارکننده P16 در سرطانهای انسانی است و ازآنپس، تهیه فهرست ژنهای کاندید با فرض بر متیلاسیون نابجا در جزایر CpG آنها آغاز شد. در سلولهای ترانسفورم شده و سلولهای بدخیم، جزایر CpG معینی از ژنهای مهارکننده تومور خاص، هیپرمتیله میشوند، از طرفی برخلاف ظهور ناگهانی موتاسیون های ژنتیکی، احتمالا روند متیلاسیون DNA یک فرآیند تدریجی است. دو تئوری مبهم در متیلاسیون از نو پدید نابجا قابل فرضی است: نخست اینکه متیلاسیون DNA منجر به ایجاد سرطان، از مراکز متیلاسیون نرمال در مجاورت جزایر CpG خالی از متیلاسیون (برای مثال توالی Alu) منتشر میشود. تئوری دیگر وجود بذر متیلاسیون است که با متیلاسیون دی نوکلئوتیدهای CpG منفرد معینی، بروز میزان بیشتری از متیلاسیون را به آن ناحیه القاء مینماید. این فرآیند تا انجام هیپرمتیلاسیون متراکم اثر تعاونی مثبت دارد.
آنجائی که هر ژن مهارکننده تومور دور الل دارد، لذا بر اساس مدل دو ضربهای کلاسیک ندسون ( The Classic TWO-hit model of Knudson)، بایستی هر دو الل پیش از شکل گیری غیر وراثتی (Sporadic cancer) فقدان فعالیت ژن مهارکننده تومور هنگامی قسمتی از کروموزوم حاوی کپی دیگر ژن (دومین ضربه) است، لکن در اکثر موارد هر دو الل با متیلاسیون DNA غیرفعال میشوند.
در سرطانهای وراثتی (Inherited Cancers)، وقتی یک الل از یک ژن مهارکننده تومور در سلولهای زایشی دچار موتاسيون شود، فقدان دومین الل در تومور حتی با رخداد حذف کروموزمی به وقوع میپیوندد. مجدداً، متیلاسیون نواحی غنی از CpG موجود در پروموتر، احتمالا مهیا کننده دومین ضربه در سرطانهای وراثتی است. در این مورد الل فاقد موتاسیون بهطور غیر نرمال متیله شده، ولی الل موتاسیون یافته متیله نمیشود.
بنا بر دلایل ذکرشده در فوق بررسی وضعیت و تغییرات متیلاسیون نقاط CpG و مخصوصاً در جزایر CpG میتواند آشکار کننده روشهای تغییر بیان ژنها در فنوتیپ های مشاهده میباشد.
تأثیر تیمار با بیسولفیت بر روی توالی DNA
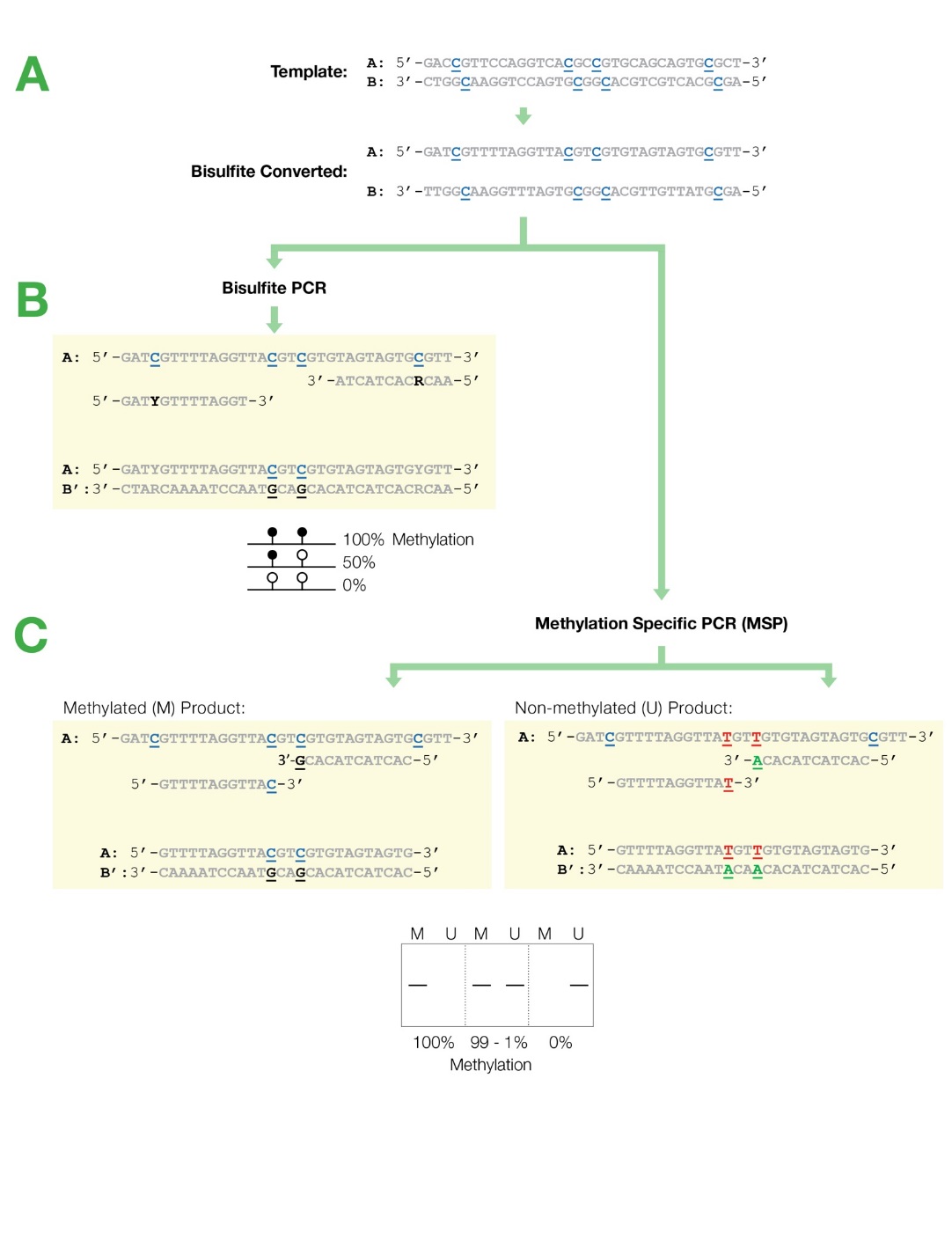
تیمار DNA با بیسولفیت ( HSO3) سبب میشود که بازهای غیر متیله سیتوزین گروه آمین خود را از دست بدهد و به باز یوراسیل تبدیل شود که همانند باز تیمین مکمل باز آدنین میباشد بنابراین در واقع باز سیتوزین بدون متیله به باز T تبدیل خواهد شد (شکل زیر قسمت A) اما درصورتیکه باز سیتوزین بهصورت متیله باشد تیمار بیسولفیت اثری بر روی این باز نخواهد داشت (شکل زیر قسمت B). بنابراین بهوسیله تیمار DNA بهوسیله بیسولفیت و بررسی تبدیل باز C به باز T میتوان به متیله بودن و یا غیر متیله بودن بازهای C پی برد.
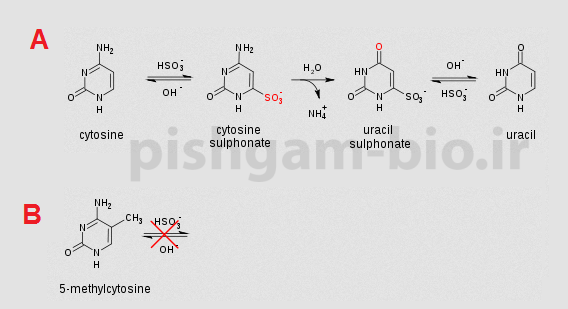
شکل فوق تأثیر تیمار DNA را با بیسولفیت بر روی بازهای سیتوزین متیله و غیر متیله نشان میدهد.
در واکنش تیمار DNA با بیسولفیت، ابتدا DNA برای ایجاد رشتههای تکی دناتوره میشود و سپس با سدیم بیسولفیت تیمار شده تا نوکلئوتیدهای سیتوزین غیر متیله به یوراسیل تبدیل شوند درحالیکه نوکلئوتیدهای متیله سیتوزین (5mC) دست نخورده باقی میمانند.
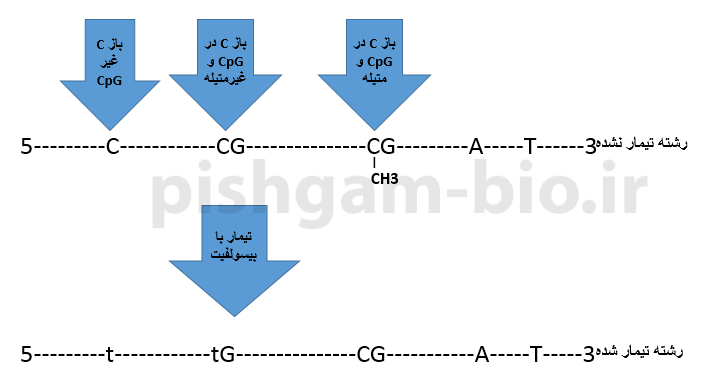
شکل فوق تأثیر تیمار بیسولفیت را بر روی بازهای DNA و همچنین سیتوزین متیله نشان میدهد. همانگونه که مشاهده میگردد تمام بازهای C غیر CpG و همچنین بازهای C در ناحیه CpG که متیله نمیباشند به باز t تبدیل میشوند.
نکته بسیار مهم: در این آموزش بازهای C که در اثر تیمار با بیسولفیت به باز تیمیدین تبدیل میشوند را با t و بازهای تیمیدین اصلی توالی را با T نشان میدهیم. بنابراین t بیانگر بازهای C غیر متیله میباشد که در اثر تیمار با بیسولفیت به باز تیمیدین تبدیل شدهاند.
توجه نمایید که دو رشته DNA بعد از تیمار با بیسولفیت دیگر با یکدیگر مکمل نخواهند بود به شکل زیر توجه نمایید.
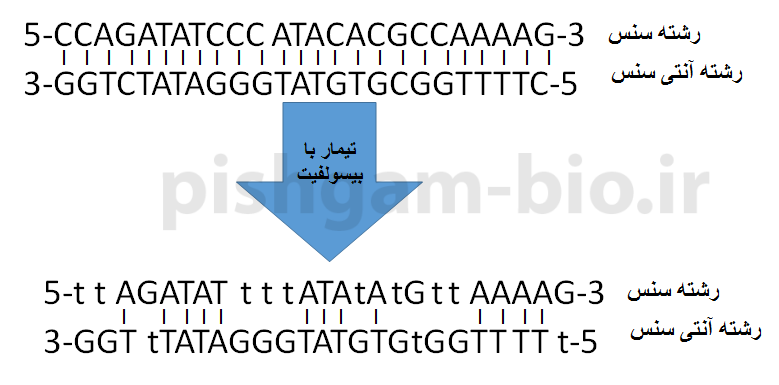
شکل فوق بیانگر تغییر توالی دو رشته DNA بعد از تیمار با بیسولفیت میباشد، همانگونه که مشاهده میگردد بعد از تیمار با بیسولفیت دو رشته DNA دیگر با یکدیگر مکمل نخواهند بود، بنابراین بعد از تیمار با بیسولفیت باید انتخاب نمود که بررسی متیلاسیون بر روی رشته سنس و یا آنتی سنس بررسی گردد. با اینکه معمولاً متیلاسیون بر روی رشته سنس بررسی میگردد اما با بررسی مقالات باید این موضوع برای ژن موردنظر اثبات شود.
بعد از تیمار رشتههای DNA با بیسولفیت نوبت بررسی تغییر بازهای C به t با روش PCR میباشد. برای بررسی متیلاسیون توسط روش بیسولفیت دو روش اصلی بر اساس تکنیک PCR وجود دارد. روش methylation-Specific PCR (MSP) و روش bisulfite Sequencing PCR(BSP) شایعترین روشهای استفاده شده برای تشخیص متیلاسیون میباشند. که در ادامه این روشها و نحوه طراحی پرایمر برای این دو روش را توضیح خواهیم داد.
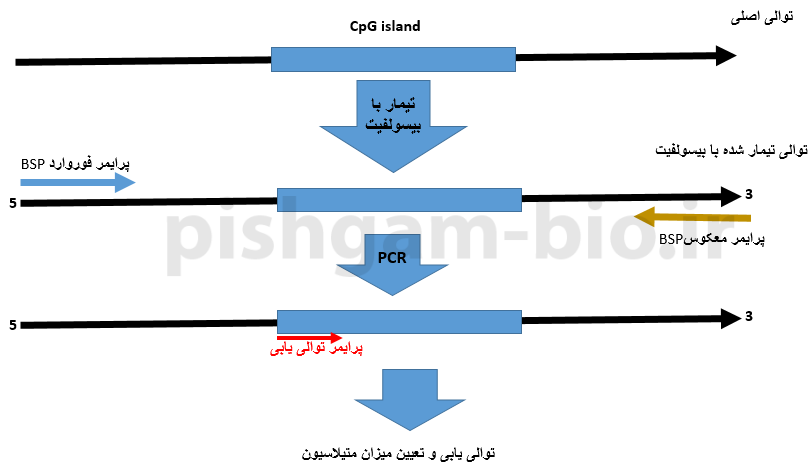
آموزش روش bisulfite Sequencing PCR(BSP) در بررسی متیلاسیون DNA
روش BSP مبتنی بر توالی یابی قطعه موردبررسی میباشد. در این روش ابتدا توالی DNA استخراج شده از نمونهها بهوسیله بیسولفیت تیمار میگردد و سپس برای دو طرف جزیره CpG پرایمر طراحی شده و سپس قطعه تکثیر شده تعیین توالی میگردد.
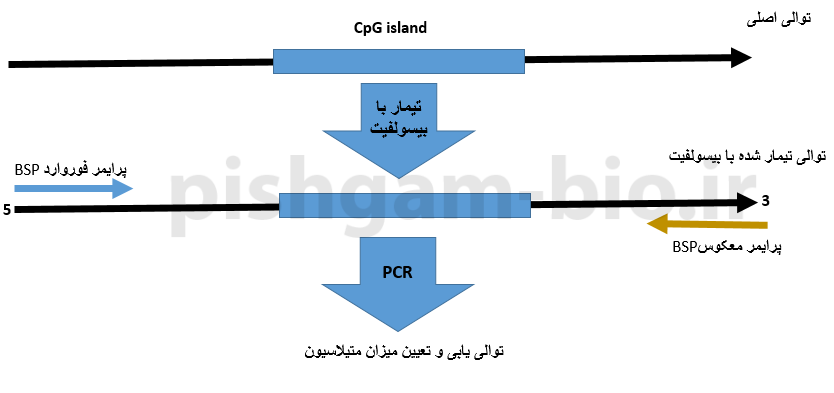
در شکل فوق طراحی پرایمر برای روش bisulfite Sequencing PCR(BSP) را نشان میدهد. قبل از توضیح سایر قوانین مربوط به طراحی پرایمر برای روش BSP این نکته را متذکر میکنیم که توالی پرایمر فوروارد بخشی از توالی قطعه موردبررسی میباشد و با آن هم جهت میباشد، اما توالی پرایمر معکوس (reverse) بهصورت reverse-complement با توالی قطعه موردنظر میباشد به شکل زیر توجه نمایید.

شکل فوق نشان دهنده جهت پرایمرهای فوروارد و معکوس و همچنین توالی موردبررسی میباشد. جهت پرایمر فوروارد هم جهت با توالی موردبررسی میباشد اما جهت پرایمر معکوس بهصورت معکوس با توالی موردبررسی میباشد. توجه داشته باشید که سمت 3پریم پرایمرها در هنگام طراحی دارای اهمیت فراوانی میباشد.
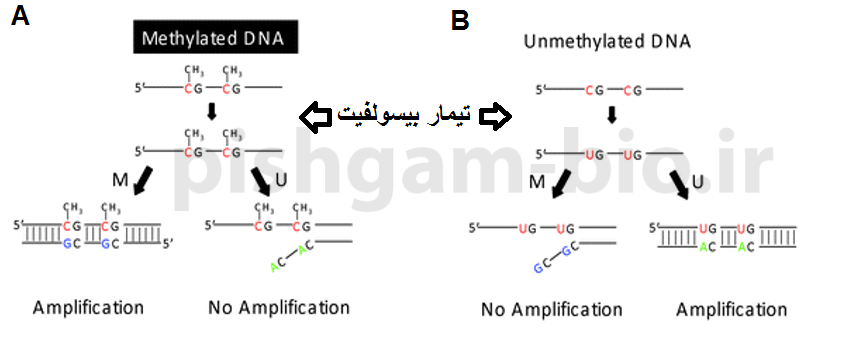
آموزش روش MSP در بررسی متیلاسیون ژنها
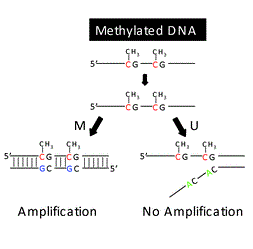
روش MSP نیازمند دو جفت پرایمر میباشد، یکی جفت پرایمر فوروارد و معکوس اختصاصی برای DNA تیمار شده با بی سولفیت نوع متیله ( جفت M) و یک جفت پرایمر فوروارد و معکوس اختصاصی برای DNA تیمار شده با بیسولفیت از نوع غیرمتیله (جفت U). بنابراین برای هر نمونه دواکنش PCR با هر جفت پرایمر انجام میشود. تکثیر با جفت پرایمرهای M متیلاسیون جایگاههای CpG را نشان میدهد (شکل زیر قسمت A) اما تکثیر با جفت پرایمرهای U بیانگر عدم متیلاسیون در جایگاههای پرایمر میباشد(شکل زیر قسمت B). یک احتمال دیگر هم وجود دارد و آن هم این است که توالی توسط هر دو جفت پرایمر M و U تکثیر شود که این بیانگر متیلاسیون جزئی این توالی میباشد.
روش Methylight BSP در بررسی متیلاسیون
طراحی پرایمر برای روش Methylight BSP شبیه به طراحی پرایمر برای BSP معمولی میباشد. اما در این روش به جای توالی یابی از دو پروب برای سنجش کمی تغییرات متیلاسیون استفاده میگردد.
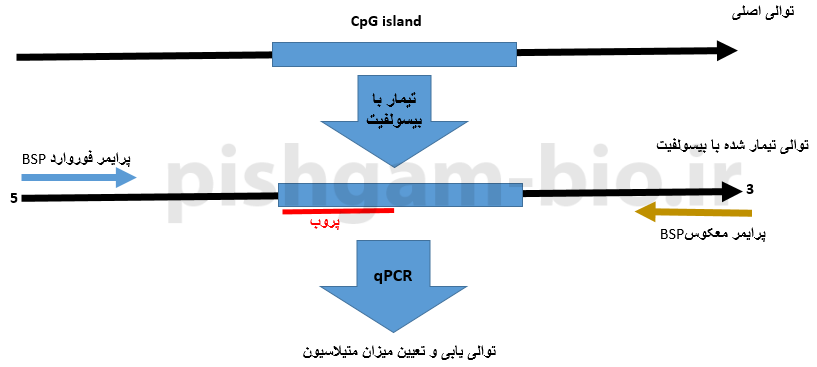
شکل فوق طراحی پرایمر و پروب در روش Methylight BSP را نشان میدهد. وضعیت متیله بودن CpGها باید توسط پروب های اختصاصی توالی متیله و توالی غیر متیله مشخص شود. در این گزینه به دو پرایمر فوروارد و معکوس و دو پروب نیاز است. پروب باید جایگاهی طراحی شود که جزایر CpG قرارگرفته است. در واقع یکی از پروب ها باید به توالی متیله و پروب دیگر باید به توالی غیرمتیله متصل شود.
روش Nested BSP در بررسی متیلاسیون
طراحی پرایمر در این گزینه شبیه طراحی پرایمر برای BSP معمولی میباشد با این تفاوت که در این روش علاوه بر دو پرایمر BSP معمولی، دو پرایمر داخلیتر نیز طراحی میشود که سبب میشود قطعه موردبررسی با اختصاصیت بالاتری تکثیر شود.
در این روش برای دو طرف مناطق CpG دو جفت پرایمر خارجی و پرایمر داخلی طراحی میشود و بعد از تکثیر توالی توسط پرایمر داخلی، توالی مربوطه توالی یابی میشود.
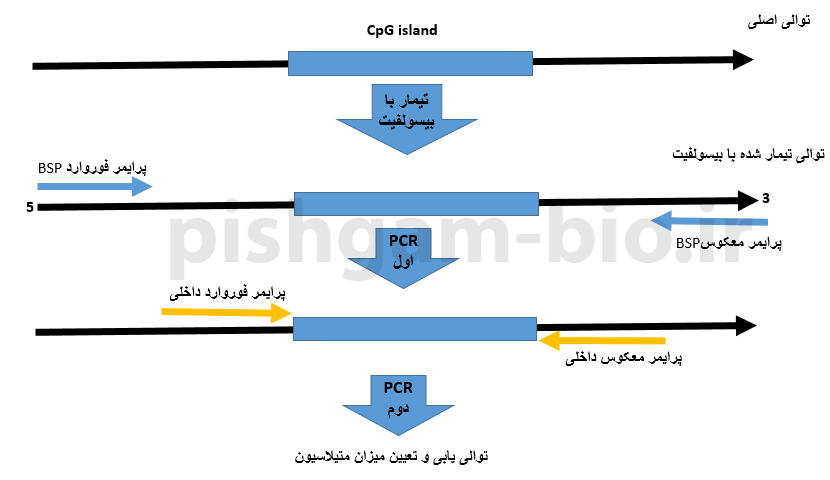
شکل فوق طراحی پرایمر در روش Nested BSP را نشان میدهد. در این گزینه به دو پرایمر فوروارد و معکوس خارجی و یک مجموعه پرایمر فوروارد و معکوس داخلی وجود دارد.
آموزش روش CORBRA در بررسی متیلاسیون
طراحی پرایمر در این گزینه شبیه نیز طراحی پرایمر برای BSP معمولی میباشد با این تفاوت که در این روش تشخیص متیلاسیون توسط آنزیمهای محدودگر صورت میپذیرد.
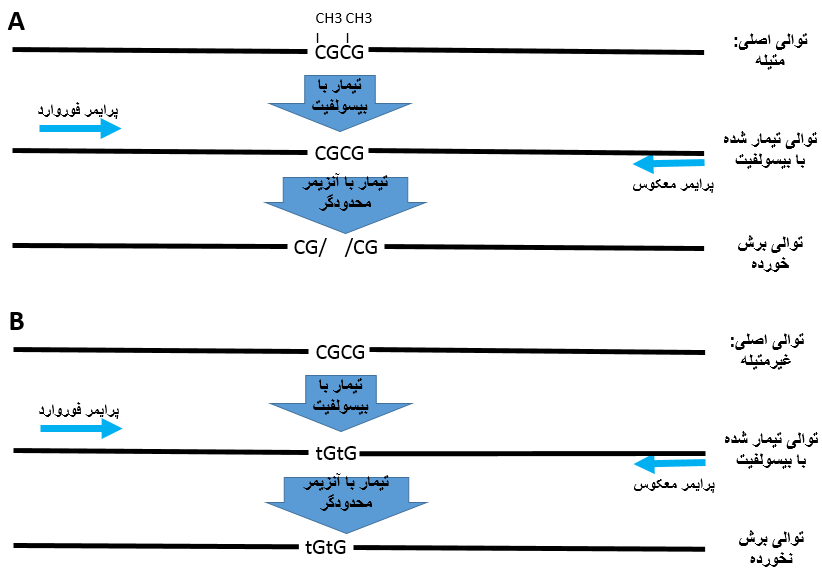
در این روش برای دو طرف منطقه CpG یک جفت پرایمر طراحی میشود و بعد از تکثیر توالی توسط پرایمرها، با آنزیم محدودگر تیمار میشود. تبدیل بازهای C در مناطق CpG میتواند سبب از بین رفتن یک جایگاه شناسایی و یا برعکس ایجاد یک جایگاه شناسایی گردد و از این طریق میتوان متیله بودن و یا متیله نبودن یک قطعه را مشخص نمود.
باید به این نکته اشاره کنیم که درصورتیکه برای توالی موردبررسی آنزیم محدودگری یافت نشود نمیتوان از این روش استفاده نمود.
آموزش روش Methylight MSP در بررسی متیلاسیون
طراحی پرایمر برای روش Methylight MSP بسیار شبیه روش MSP معمولی میباشد. در این روش نیز ما یک جفت پرایمر فوروارد و معکوس مختص توالی متیله و یک جفت پرایمر فوروارد و معکوس مختص توالی غیر متیله داریم. اما علاوه بر دو جفت پرایمر فوق یک پروب نیز باید طراحی شود.
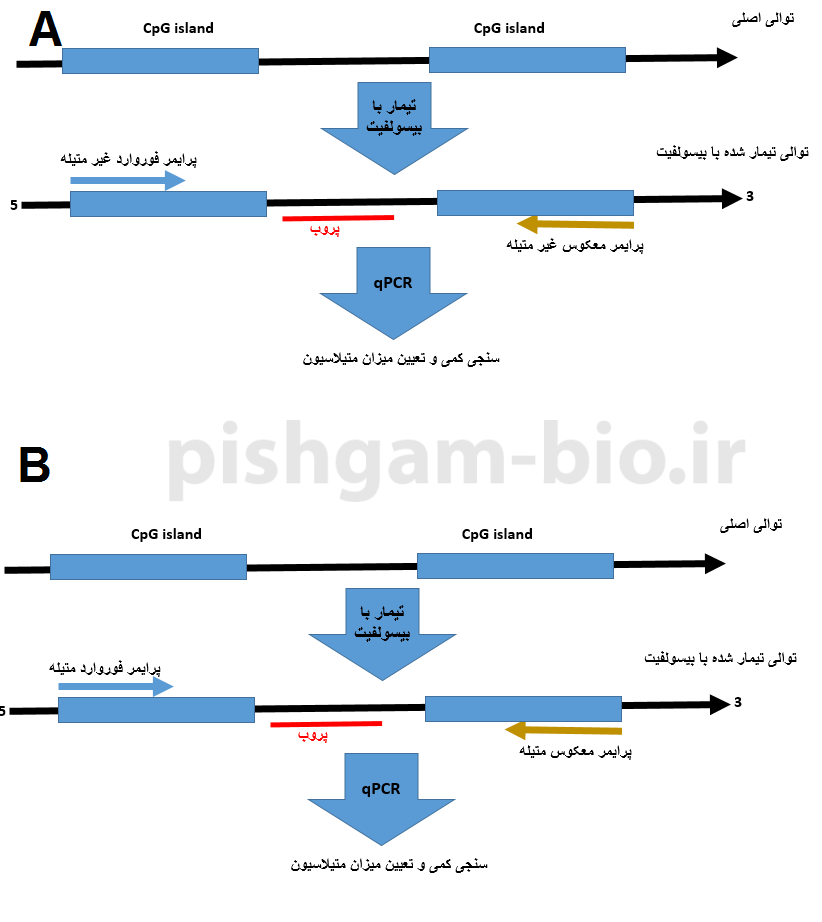
شکل فوق اصول روش Methylight MSP را نشان میدهد.
نکته: به دلیل اینکه در این روش از پروب استفاده میشود میتوان میزان متیلاسیون را بهصورت کمی با روش ریل تایم موردبررسی قرار داد.
آموزش روش Nested MSP در بررسی متیلاسیون ژنها
با انتخاب گزینه Pick Nested MSP primers میتوان برای روش Nested MSP طراحی پرایمر نمود. طراحی پرایمر برای Nested MSP شامل 3 جفت پرایمر میباشد. جفت پرایمر اول که بهعنوان پرایمر فوروارد و معکوس خارجی شناخته میشود شبیه پرایمرها در روش BSP معمولی میباشد. به این صورت که این جفت پرایمر باید بین توالی DNA تیمار شده با بیسولفیت و توالی تیمار نشده تمایز قائل شود، اما بین توالی متیله و غیر متیله تمایزی قائل نشود. جفت پرایمر دوم پرایمر فوروارد و معکوس داخلی نوع M (متیله) میباشد و جفت پرایمر سوم، پرایمر فوروارد و معکوس داخلی نوع U (غیر متیله) میباشد.
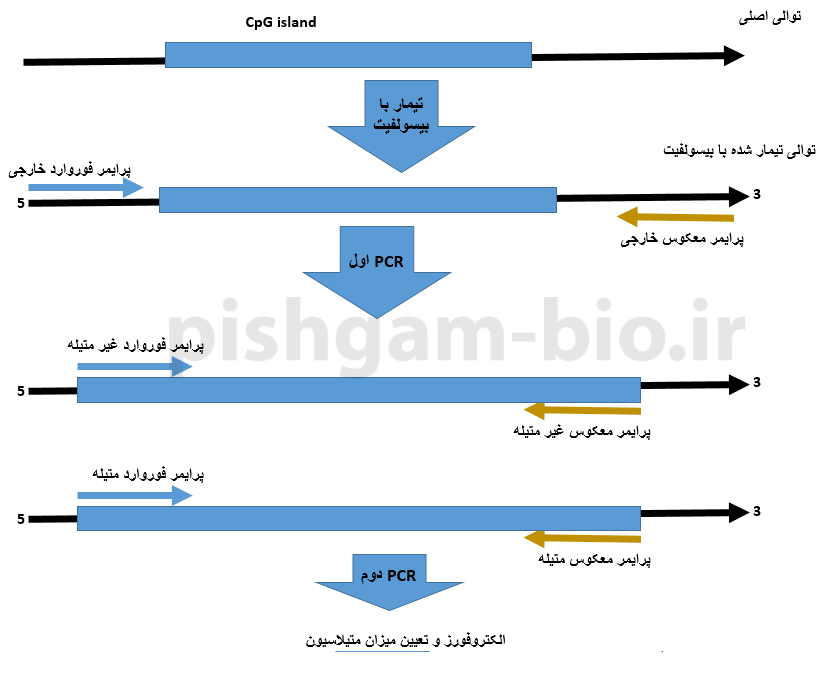
شکل فوق روش Nested MSP را نشان میدهد. در مرحله اول PCR با استفاده از پرایمرهای فوروارد و معکوس خارجی انجام میپذیرد.
مرحله دوم این روش PCR با استفاده از پرایمرهای فوروارد و معکوس داخلی نوع M و U میباشد. که در این مرحله متیله بودن و یا غیرمتیله بودن توالی مشخص خواهد شد.
آموزش روش SNuPE برای بررسی متیلاسیون
SNuPE مخفف Bisulfite SNuPE (single nucleotide primer extension) میباشد. شکل زیر اصول این روش را نشان میدهد.
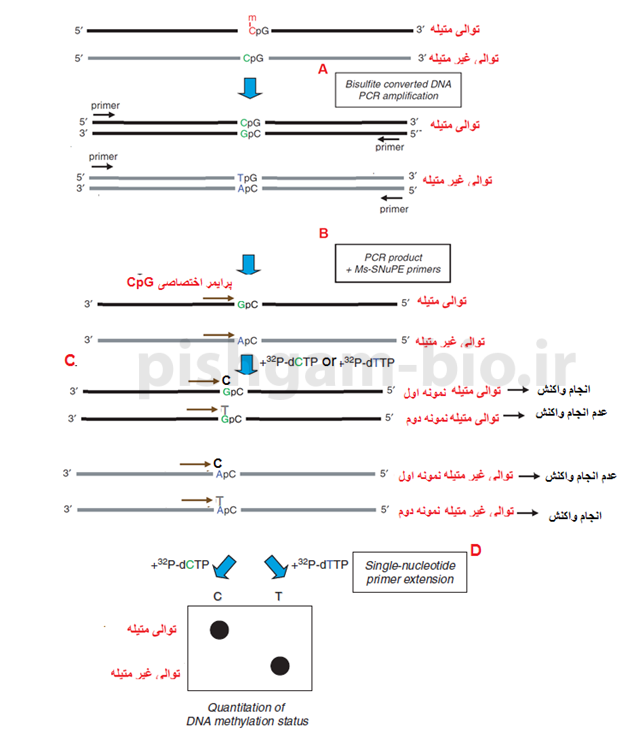
شکل M8: روشSNuPE برای بررسی متیلاسیون در یک جایگاه CpG میباشد و شامل 4 مرحله میباشد. در مرحله A ما دو نمونه DNA داریم که یکی از نمونهها متیله و دیگری غیر متیله میباشد. و در مرحله اول هر دو نمونهها بهوسیله بیسولفیت تیمار خواهند شد. و سپس بهوسیله پرایمرها دو نمونه فوق تکثیر خواهند شد.
در مرحله B محصول PCR مرحله قبل با پرایمر اختصاصی جایگاه CpG مخلوط میشود.
در مرحله C هر یک از نمونههای مرحله قبل به دو قسمت تقسیم میشوند و به قسمت اول هر نمونه باز dCTP نشاندار و به قسمت دوم هر نمونه باز dTTP نشاندار اضافه مینمایند. همانگونه که مشاهده مینمایید باز dCTP تنها به نمونه متیله و باز dTTP تنها به نمونه غیر متیله متصل میشود.
از آنجایی که دو باز فوق بهصورت نشاندار میباشند بعد از اتصال دو باز به پرایمر اختصاصی ناحیه CpG میتوان در مرحله D این پرایمرهای نشاندار را موردبررسی قرار داد و متیله بودن توالیها را موردبررسی قرار داد.
روش Bisulfite pyrosequencing در بررسی متیلاسیون
در روش Bisulfite pyrosequencing متیلاسیون چند جایگاه CpG بسیار نزدیک به هم توسط روش توالی یابی pyrosequencing مشخص میشود. در این تکنیک نیز در مرحله اول توالی DNA تیمار شده با بیسولفیت توسط دو پرایمر فوروارد و معکوس BSP تکثیر میگردد. سپس به دلیل اینکه در PCR محصولات دو رشتهای هستند یکی از رشتهها جداسازی و در مرحله دوم توالی یابی میشود. اما تولی یابی به روش pyrosequencing انجام میپذیرد که توالی یابی به روش pyrosequencing نیازمند یک پرایمر جداگانه میباشد.
شکل M12: شکل فوق دو مرحله بررسی متیلاسیون به روش pyrosequencing را نشان میدهد. به دلیل اینکه در pyrosequencing تعداد بازهای خوانش شده بسیار کوتاه میباشد (نهایتاً تا 30 باز، که بستگی به نوع دستگاه هم دارد) تعدا جایگاه های CpG مورد بررسی نیز محدود می باشد.
پایان بخش مقدماتی
در بخش های فوق، روشهای بررسی متیلاسیون مبتنی بر طراحی پرایمر را به اختصار توضیح دادیم. فایل آموزش پیشرفته طراحی پرایمر برای متیلاسیون شامل آموزش پیشرفته تر تکنیکهای فوق به همراه نکات اختصاصی در طراحی پرایمر برای هر تکنیک می باشد و در ادامه نحوه طراحی پرایمر برای تکنیکهای فوق با نرم افزارهای مختلف آموزش داده شده و سپس صحت پرایمرهای طراحی شده توسط PCR مجازی توضیح داده شده است. می توانید لیست کامل آموزش های فایل پیشرفته را در فهرست ارائه شده در ابتدای همین آموزش مشاهده نمایید.
برای تهیه آموزش پیشرفته طراحی پرایمر برای متیلاسیون از لینک زیر اقدام نمایید.










دیدگاهتان را بنویسید