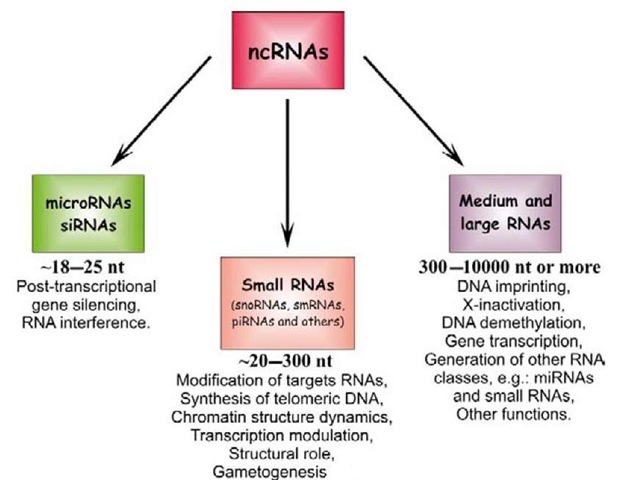
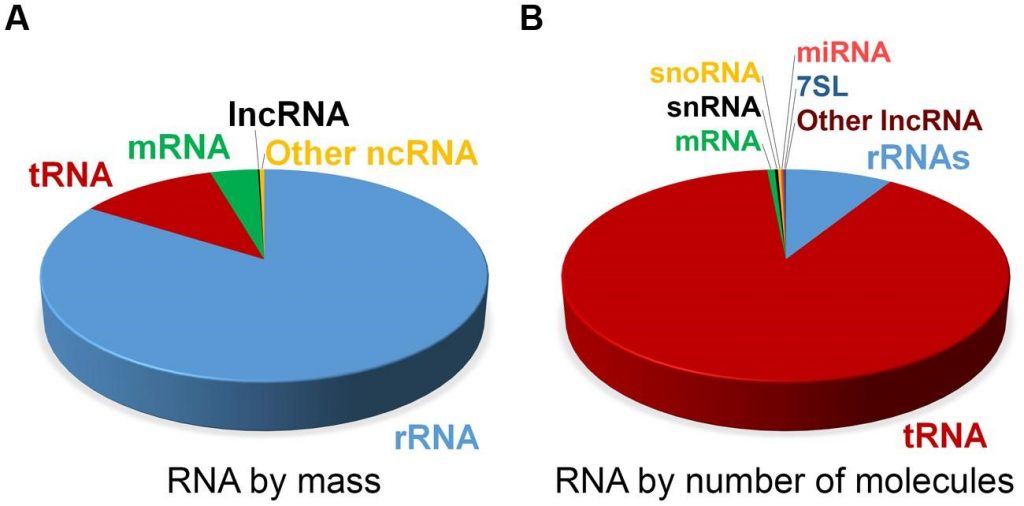
دیدگاه جدید در مورد ترانسکریپتوم پستانداران یک الگوی پیچیدهای از رونوشتهای کدکننده[1] و غیرکدکننده[2] پروتئین که به صورت همپوشان و غیر همپوشان در ژنوم قرار دارند را نشان میدهد. جدیدا مشخص شده است که نواحی غیر کد کننده یا RNAهای غیر کد کننده[3] پروتئین نقش اساسی در مکانیسمهای مختلف تنظیم بیان ژنها بازی میکند. استفاده از روشهایی که بر مبنای توالییابی نسل دوم DNA استوارند (مثل RNA-seq) و نیز آنالیز ترانسکریپتوم سراسری[4] و نیز روشهای tiling array نشان داده اند که ژنوم یوکاریوتها، بالغ بر 90% به ترانسکریپتوم رونویسی میشوند که این بیان کننده وجود مقدار عظیمی RNA غیر کد کننده (ncRNA) در سلول میباشد. همچنین این روشها و تکنولوژیهای نوین نشان دادهاند که ژنوم یوکاریوتها به واسطه تغییرات بزرگ ساختاری مثل حذفهای بزرگ و درج شدگی های بزرگ[5] بسیار پویا میباشد]8[.
بیش از یک دهه گذشته بر اساس اطلاعات حاصل از توالی یابی ESTها و دیگر تکنولوژیهای آن زمان تصور بر این بود که ژنوم انسان شامل حدود 20000 تا 30000 ژن میباشد اما در حال حاضر با گشوده شدن پنجره جدیدی از ترانسکریپتوم به نام ncRNA این تخمین از ژنهای انسانی به طرز قابل توجهی تغییر خواهد یافت. ارگانیسمهایی مثل کرم الگانس[6] از لحاظ تعداد ژنهای کد کننده پروتئین تقریبا همانند انسان می باشد و فقط اختلاف اندکی دارد که این اختلاف اندک نمیتواند توجیه کننده پیچیدگی عظیم بدن انسان در مقابل کرم الگانس باشد بنابراین میتوان گفت که وجود گروه ncRNAها میتواند باعث بروز پیچیدگیهای بیشتر در ژنوم انسان شده است]9[.
در گذشته محققان بر این باور بودند که قسمت کوچکی از ژنوم یوکاریوتها به پروتئین کد میشود (حدود 2%) و قسمت کوچک دیگری هم به RNAهای ریبوزومی[7] و RNAهای انتقالی[8] تبدیل میشود و بقیه قسمتهای ژنوم زاید میباشد. اما جدیدا ncRNAها طویل یا بزرگ[9] که در تغییرات کروماتین دخالت دارند و RNAهای کوچکی که در نواحی پروموتر[10] نسخه برداری میشوند و نیز RNAهایی که از نواحی تکراری نسخه برداری میشوند و همچنین ncRNAهای میتوکندریایی نیز گزارش شدهاند]10[.
اهمیت عملکرد آرناهای غیر کد کننده ( non coding RNAیا ncRNAها) در پدیده های زیستی
اهمیت ncRNAها وقتی بیشتر شد که کشفی در ارتباط با دخالت ncRNAها در انتقال اطلاعات اپیژنتیکی از نسلی به نسل بعد در گیاهان و موش گزارش شد ]11،12[. اگرچه پدیده اپیژنتیک نسلهای مختلف[11] مبحث نوینی نیست و در گذشته مکانیسمهایی از قبیل تغییرات DNA و مارکرهای هیستونی در این باره گزارش شده بودند اما این کشفیات جدید دخالت ncRNAها را در فصل جدیدی از قوانین وراثت نشان میدهد. و نیز با برخی از جنبههای نظریه لامارک سازگار است و از آن به عنوان لامارکیسم نرم[12] یاد میشود. اثرات اپی ژنتیکی نسلهای متفاوت پدیدهی پیچیدهای میباشد که ممکن است به واسطه RNAها کنترل شود. یک مقاله کلیدی توسط Hejimans و همکاران در سال 2008 منتشر شد که اثبات کننده این مسئله میباشد که شرایط محیطی روزهای اولیه زندگی (رحم مادر) میتواند سبب تغییرات اپیژنتیکی پایهای در انسان بشود و این عوامل ایجاد تغییرات اپیژنتیکی میتوانند ncRNAها باشند فلذا این مقاله اهمیت فرضیه دخالت RNA را در اپیژنتیک نسلها متفاوت اثبات میکند ]12[.
نقش Deep Sequencing (مثل RNA-seq) در شناسایی آرناهای غیر کد کننده ( non coding RNAیا ncRNAها) و میکروارناها
اطلاعات حاصل از Deep Sequencing جدیدا نشان داده است که ncRNAهای طویل و کوتاه از نظر تعداد بسیار بیشتر از RNAهای کد کنندهی پروتئین میباشند فلذا اثبات کنندهی این مسئله میباشد که ژنوم انسان برخلاف دیدگاه سابق از لحاظ نسخه برداری بسیار فعالتر می باشد]13[.
تکنولوژیهای نوین توالی یابی DNA باعث فراهم شدن امکان دسترسی دقیقتر ترانسکریپتهای یوکاریوتها شده است]14[. مطالعات متفاوت که مستقل از همدیگر از رویکردهای متفاوتی جهت بررسی ترانسکریپتوم استفاده کرده بودند به این نتیجه رسیدهاند که مقدار مادهی ژنتیکی رونویسی شونده موجود در سلولهای یوکاریوتی بسیار بیشتر از آن چیزی است که در گذشته تصور میکردهاند ]15تا18[. این یافتههای جدید در مقابل دیدگاههای گذشته قرار میگیرد آنجا که میگفتند که نواحی غیر کد کننده ژنوم انسان نواحی زاید ساختاری هستند و یا به عنوان نواحی تنظیمی بیان ژنوم (یعنی افزایندهها[13] و خاموش کنندهها[14]) میباشند. این مسئله که نواحی تنظیمی ژنوم مثل نواحی افزاینده می توانند رونویسی بشوند بسیار فریبنده است]19[. هنوز بر عملکرد تمام این ncRNAها اتفاق نظر وجود ندارد مخصوصا به این خاطر که بیشتر آنها به مقدار خیلی کم بیان میشوند]20[. اما باید توجه نمود که بیان کم این RNAها دلیل بر عدم وجود عملکرد برای آنها نیست چرا که عملکرد یک سری از آنها در فرایندهای اپیژنتیکی گزارش شده است]21[.
مطالعات جدید طی بررسی ترانسکریپتوم مخمر مشخص نموده است که قسمت اعظم ژنوم مخمر که توالیهای غیر کد کننده نامیده شده بودند به RNA رونویسی میشوند. ژنوم مخمر تحت شرایط مختلف مورد بررسی قرار گرفت و مشخص شد که ژنوم مخمر یک انعطاف (remodeling) خاصی طی شرایط مختلف نشان میدهد و بیش از 90% ژنوم آن رونویسی میشود]22،17[. دو گروه تحقیقاتی که به طور مجزا کار میکردند با استفاده از رویکرد tiling array اقدام به بررسی سطح بیانی و ساختار رونوشتهای مخمر نمودهاند. چندین رونوشت جدید گزارش شد که بسته به کاری که در سلول انجام میدهند نامگذاری شدند. برای مثال SUTها[15] که عمر زیادی در سلول دارند همراه با CUTها[16] که خیلی سریع به واسطه مکانیسم RNAی مراقب[17] تخریب میشوند، شناسایی شدند. یکی از مشخصههای بارز این نوع از RNAها (CUTs ، SUTs) این است که در ژنوم با همدیگر همپوشان می باشند و این ویژگی آنها، این سوال را در ذهن ایجاد میکند که چگونه بیان این RNAها تنظیم میشود. کشف دیگری که از این مطالعات حاصل شد این بود که این RNAها اغلب در ارتباط با قسمتهای کد کننده ژنوم میباشند و پروموترهای دو جهتی[18] دارند]23،24[.
نقش پروژه ENCODE در شناسایی آرناهای غیر کد کننده ( non coding RNA یا ncRNAها) و میکروارناها
در ارتباط با سلولهای انسانی، پروژه ENCODE[19] قادر بود که رونوشتهای مختلف سلولهای انسانی را با رویکرد Deep sequencing آنالیز کند. با این رویکرد RNAهای کوچک (کمتر از 200 نوکلئوتید) با برچسبهای CAGE[20] که در سر 5 پرایم RNAهای بزرگ شناخته شده قرار دارند همردیف[21] شدند. RNAهای کوچک مربوط به پروموتر (PASR)[22] و RNAهای بزرگ مربوط به پروموتر (PALR) نیز شناسایی شدند. PASRها حدود 20% از کل RNAهای شناخته شده را شامل میشوند و در نواحی ژنی و بین ژنی قرار دارند]25[. تحقیقات دیگری با استفاده از رویکردهای مشابه ثابت نمودند که ترانسکریپتهایی از نواحی تکراری ژنوم منشا میگیرند (پروژه CAGE مربوط به FANTOM[23]). حدود نصف ژنوم انسان توالیهای تکراری هستند و شامل عناصر رتروترانسپوزونی مثل LINE و SINE[24] میباشند. تحقیقی که Fanlker و همکاران انجام دادند مشخص نمود که در انسان 30% از ترانسکریپتها از نواحی تکراری منشا میگیرند و بیشتر آنها ncRNA هستند؛ در نتیجه این تحقیق مشخص میکند که عناصر رتروترانسپوزونی نقش اساسی در بازدهی ژنوم انسانی دارند]26[. گروههای تحقیقاتی دیگری نیز نشان دادند که توالیهای Alu و SINE در سلولهای مختلف انسان قادرند بیان شوند. این مطالعات جز اولین مطالعاتی بودند که اثبات کردند عناصر تکراری در ژنوم انسان بیان میشوند]26،27[.
روی همرفته مطالعات اخیر نشان داده است که ncRNAها نقش اساسی در ژنوم انسان دارند و بر خلاف دیدگاههای گذشته ژنوم یوکاریوتها بسیار پیچیده بوده و ژنها ممکن است به صورت همپوشان (خواه همجهت و یا آنتی سنس) روی رشتهی DNA قرار داشته باشند و عناصر تکراری و ncRNAهای زیادی با عملکرد نامشخص به واسطه مکانیسمهای مختلفی رونویسی میشوند.











دیدگاهتان را بنویسید