آشنایی با نرم افزار TargetP برای تعیین عملکرد پروتئین ها
یکی از روش های بسیار کارآمد در تعیین عملکرد پروتئین ها پیش بینی بیوانفورماتیکی محل جایگیری و فعالیت پروتئین ها در سلول ها می باشد. جایگیری پروتئین ها در میتوکندری ، کلروپلاست، سیتوپلاسم و یا هسته تا میزان زیادی تعیین کننده نقش آن پروتئین می باشد. با استفاده از نرم افزار آنلاین TargetP می توان به صورت بیوانفورماتیکی و با شناسایی توالی های سیگنال وهم چنین محل برش برای سیگنال پپتید موجود در توالی پروتئین ها محل عملکرد پروتئین ها را پیش بینی نمود.
کاربرد های نرم افزار آنلاین TargetP به شرح زیر است:
تعیین امتیاز احتمال قرار گیری پروتئین ها در کلروپلاست
تعیین امتیاز احتمال قرار گیری پروتئین ها در میتوکندری
تعیین امتیاز احتمال قرار گیری پروتئین ها در مسیرهای ترشحی
آدرس سایت TargetP:
http://www.cbs.dtu.dk/services/TargetP/
برای ورود به نرم افزار TargetP بر روی بینک فوق کلیک نمایید. تا سایت زیر در مرورگر شما باز شود.
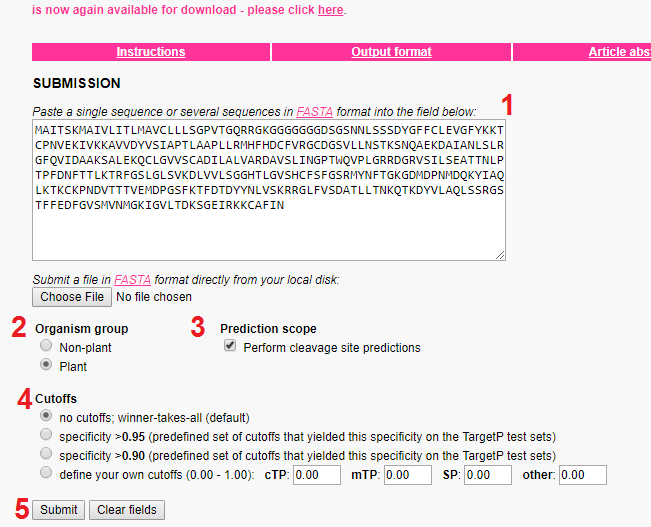
شکل فوق صفحه اصلی نرم افزار TargetP را نشان می دهد. کار با این نرم افزار بسیار ساده می باشد.
ابتدا باید توالی پروتئینی را در کادر مخصوص وارد نمایید.
در مرحله بعد باید در قسمت Organism group تعیین نمایید که توالی پروتئینی به گیاه (Plant) و یا موجودات غیر گیاهی (Non-plant) تعلق دارد.
در مرحله بعد می توانید در قسمت Prediction scope تیگ گزینه Perform cleavage site predictions را فعال نمایید. با فعال نمودن این گزینه پیش بینی وجود سیگنال پپتید از طریق شناسایی محل برش سیگنال پپتید امکان پذیر می شود.
سپس در مرحله بعد برای قسمت Cutoffs گزینه no cutoffs; winner-takes-all (default) را انتخاب نمایید تا همه نتایج بدون حذف شدن نمایش داده شوند.
سپس بر روی گزینه Submid کلیک نمایید تا فرایند اسکن توالی آغاز شود.
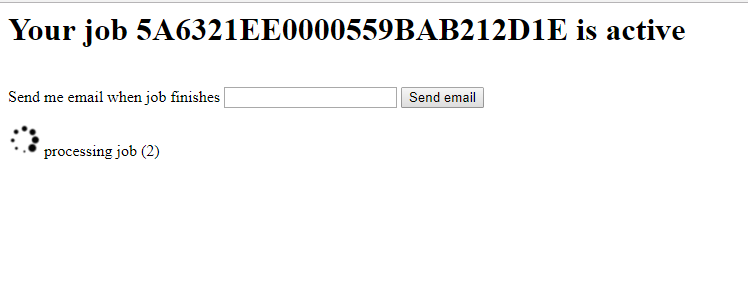
با نمایش پنجره فوق فرایند اسکن و انالیز توالی وارد شده آغاز شده است . در صورتی که این مرحله طولانی شده است می توانید در کادر مربوطه ایمیل خود را وارد نمایید تا بعد از اتمام کار نتایج به ایمیل شما ارسال شود.
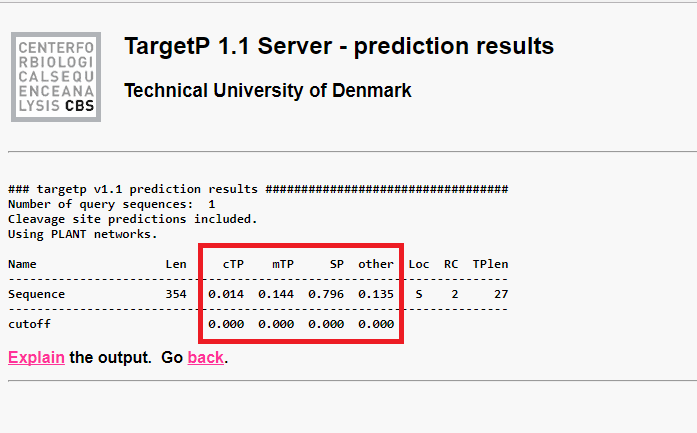
پنجره فوق نتایج آنالیز توالی پروتئینی را نشان می دهد. گزینه Len طول پروتئین وارد شده را نمایش می دهد
عدد نمایش داده شده در زیر گزینه cTP بیانگر احتمال قرار گرفتن پروتئین در کلروپلاست می باشد (chloroplast transit peptide)
عدد نمایش داده شده در زیر گزینه mTP بیانگر احتمال قرار گرفتن پروتئین در میتوکندری می باشد (mitochondrial targeting peptide)
عدد نمایش داده شده در زیر گزینه SP بیانگر احتمال وجود سیگنال پپتید (signal peptide) را در توالی پروتئینی نشان میدهد. یک پروتئین با دارا بودن سیگنال پپتید می تواند در مسیرهای ترشحی (Secretory pathway) وارد شود.
در شکل فوق امتیاز گزینه SP از سایر گزینه ها بیشتر می باشد بنابراین به احتمال فراوان پروتئین مربوطه دارای سیگنال پپتید بوده و در مسیرهای ترشحی وارد می شود.
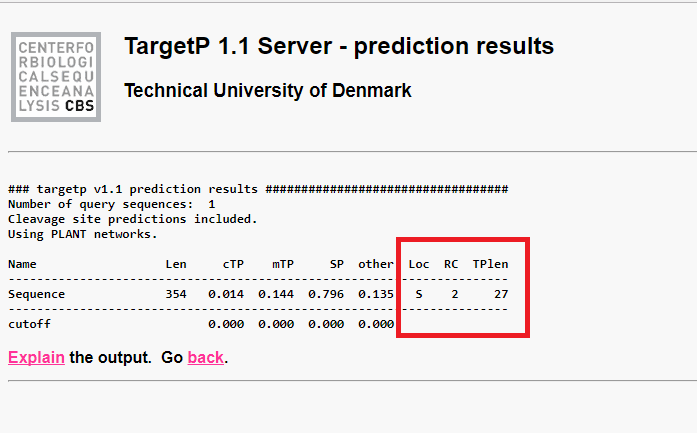
در ادامه نتایج، در زیر قسمت Loc محل جایگیری تعیین شده برای پروتئین نمایش داده می شود که تفسیر آن به شرح زیر است:
C: کلروپلاست
M: میتوکندری
S: مسیر ترشحی
عدد نمایش داده شده در قسمت RC بیانگر میزان صحت پیش بینی می باشد. این عدد بین 1 تا 5 قرار دارد که عدد به منزله پیش بینی قویتر می باشد.