در ادامه آموزش نرمافزار ORFfinder از سایت NCBI برای شناسایی نواحی ORF توالیها ارائه خواهد شد. برای یافتن نواحی ORF در نرمافزار ORFfinder میتوان از توالی RNA و یا DNA استفاده نمود.
با کلیک بر روی لینک زیر به صفحه اصلی نرمافزار ORFfinder وارد میشوید.
https://www.ncbi.nlm.nih.gov/orffinder/
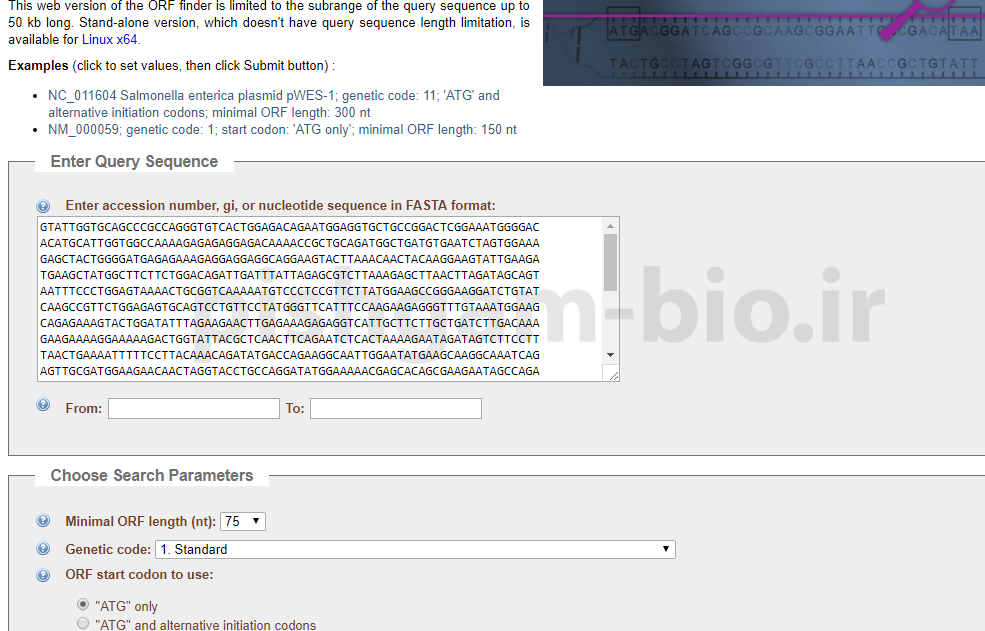
صفحه اصلی نرمافزار ORFfinder به شکل فوق میباشد.
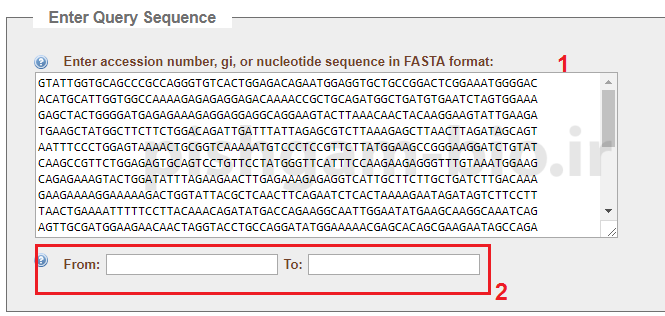
ابتدا توالی موردنظر را برای بررسی جهت یافتن ORFها در کادر مربوطه وارد نمایید. در صورتی که تمایل دارید جستجو در قسمت خاصی از توالی صورت پذیرد میتوانید در قسمتی که کادر 2 مشخص شده است ابتدا و انتهای محدوده موردنظر را وارد نمایید.
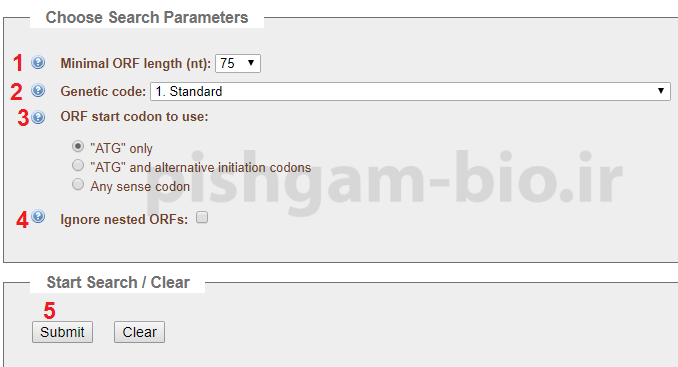
سپس:
1: در قسمت Minimal ORF length (nt): میتوانید تعیین نمایید حداقل طول ORFهایی که در قسمت نتایج نمایش داده میشود چه قدر باشد در این قسمت میتوانید بر اساس پیش بینی خود از طول پروتئینی که باید سنتز شود حداقل طول ORF را تعیین نمایید توجه داشته باشید که طول ORF سه برابر طول پروتئین سنتز شده میباشد (زیرا هر کدون ژنتیکی که کد کننده یک اسیدآمینه میباشد از سه باز تشکیل شده است).
2: در قسمت Genetic code: میتوان تعیین نمود که از کدام کد ژنتیکی برای جستجوی نواحی ORF استفاده شود. (توضیحات بیشتر در شکل بعدی)
3: در قسمت ORF start codon to use: میتوان تعیین نمود که کدون آغازین قسمت ORF رمز ATG یا رمزها جایگزین ATG باشد با انتخاب گزینه Any sense codon نرمافزار همه ORFها (چه با ATG شروع شده باشد یا خیر) را شناسایی مینماید. در این قسمت از چه گزینهای باید استفاده نمود.
برای شروع ترجمه پروتئین اولین اسیدآمینهای که در ترجمه به کار میرود متیونین (met) میباشد که کدون آن رمز ATG میباشد البته در برخی از باکتریها کدون های دیگری هم میتوانند اسیدآمینه متیونین را رمز دهی کنند. از دو گزینه “ATG” only و “ATG” and alternative initiation codons (در باکتریهایی مانند اکلی)در صورتی میتوان استفاده نمود که مطمئن باشید توالی کد کننده سمت Nترمینال پروتئین در توالی وارد شده قرار دارد (زیرا ترجمه mRNA و سنتز پروتئین از سمت Nترمینال به Cترمینال انجام میشود). اما در صورتی که اطمینان ندارید که توالی کد کنند سمت N ترمینال پروتئین در توالی وارد شده قرار دارد یا خیر (مانند مواقعی که از توالیهای partial و ناکامل استفاده میشود) باید از گزینه Any sense codon استفاده شود.
4: با زدن تیک گزینه Ignore nested ORFs: نرمافزار دیگر ORFهای کوچکی که درون ORF بزرگتر قرار گرفته است نمایش نمیدهد.
5: در آخر بر روی گزینه Submit کلیک نمایید تا جستجوی نواحی ORF آغاز شود.
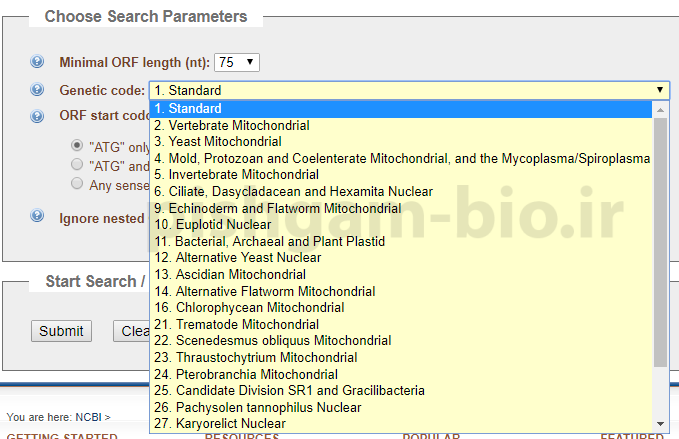
در بیشتر موجودات در فرایند ترجمه از رمزدهی ژنتیکی استاندارد استفاده میشود اما به دلیل اینکه رمزدهی ژنتیکی در تعدادی از موجودات و حتی اندامکها مانند میتوکندری مخمر و مهرهداران با رمزدهی ژنتیکی استاندارد متفاوت است باید در قسمت Genetic code: نرمافزار ORFfinder مشخص نمود که با چه رمز ژنتیکی وجود نواحی ORF در توالی وارد شده جستجو گردد. در این گزینه فهرستی از موجودات و اندامکهای مختلف که رمزدهی ژنتیکی آنها را با رمزدهی استاندارد متفاوت میباشد قابل مشاهده میباشد
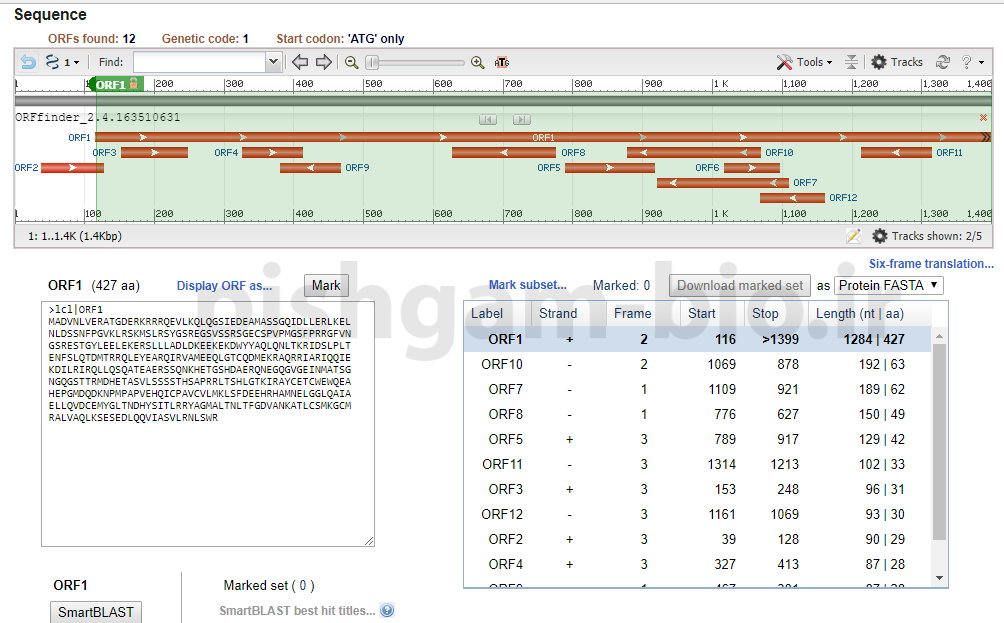
نتیجه بررسی نواحی ORF برای توالی وارد شده در پنجره فوق نمایش داده میشود.
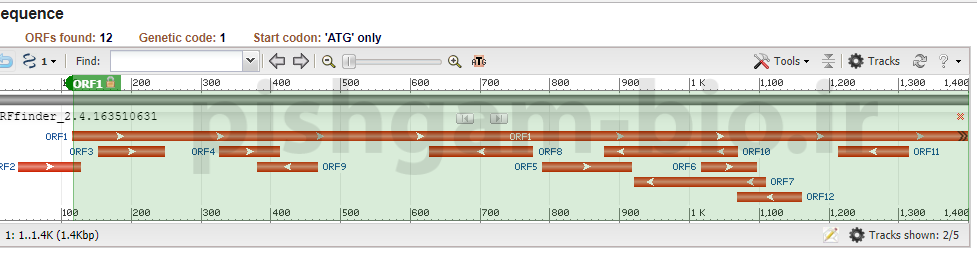
در این قسمت میتوان ORFهای شناسایی شده، طول و جهت آنها را مشاهده نمود. در قسمت ORFs found: میتوان ORFهای شناسایی شده را مشاهده نمود . با کلیک بر روی هر ORF در این قسمت، مشخصات آن در قسمتهای بعدی قابل مشاهده میباشد.
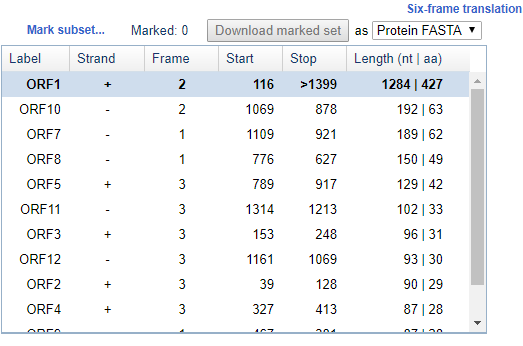
در این جدول میتوان ویژگیهای ORFهای شناسایی شده مانند طول، نقطه شروع و پایان و فریم و رشتهای که ORF در آن قرار گرفته را مشاهده نمود.
نرمافزار ORFfinder وجود ORF را در 6 فریم بررسی میکند، به دلیل اینکه هر کدون ژنتیکی از 3 عدد باز متوالی تشکیل میگردد برای هر توالی مورد بررسی میتوان 6 فریم برای خوانش و رمزدهی در نظر گرفت که 3 فریم روی رشته مثبت (همان رشتهای که وارد نمودهاید) و سه فریم روی رشته منفی ( توالی reversecomplement رشته وارد شده) قرار میگیرد. با بررسی هر شش فریم، به احتمال زیاد ORF1 باید ORF توالی وارد شده باشد زیرا دارای بلندترین طول در بین ORFها میباشد. اما توجه نمایید که در اغلب موارد وجود ORF طولانی در توالی mRNA مشاهده میشود و در صورتی که از توالی ژنومی برای جستجوی ORFها استفاده شود به دلیل وجود اینترونهای بزرگ در توالی یوکاریوتها مشاهده ORF طولانی کمتر میباشد.
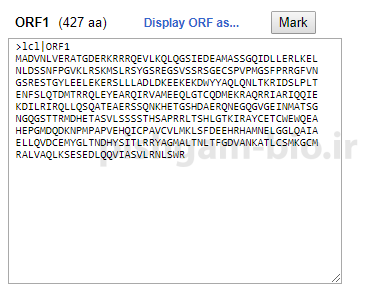
با کلیک بر روی ORF توالی پروتئینی آن در این کادر نمایش داده میشود.
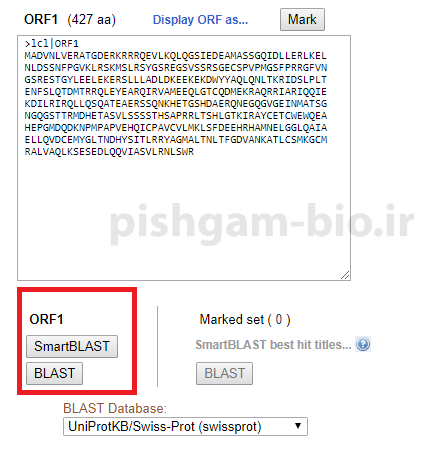
در ادامه میتوانید با استفاده از ابزار smartBLAST و یا BLAST توالی ORF ها را توالی پروتئینهای شناخته شده مورد مقایسه و هم ردیفی قرار دهید تا عملکرد پروتئین احتمالی شناسایی شده مشخص گردد.
در کانال تلگرام و سایت ما آموزش کامل بلاست ارائه شده است اما در این قسمت به این تکنیک اشاره مختصری مینماییم.
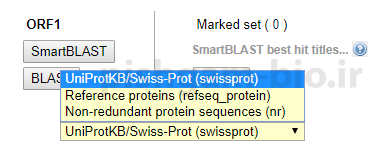
برای بلاست توالی پروتئینی از قسمت BLAST Database: یکی از پایگاههای داده پروتئینی را انتخاب نمایید. برای بررسی بهتر میتوانید بلاست را با هر سه پایگاه موجود انجام و نتایج را با ه مقایسه نمایید.
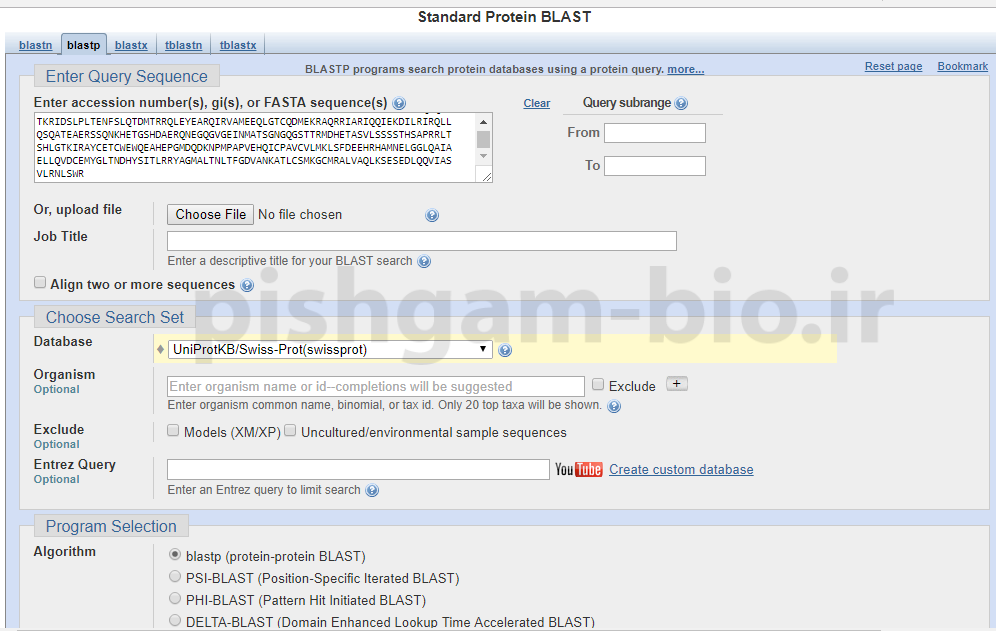
بعد از کلیک بر روی گزینههای smartBLAST و یا BLAST به صفحه اصلی بلاست پروتئین وارد میشوید.
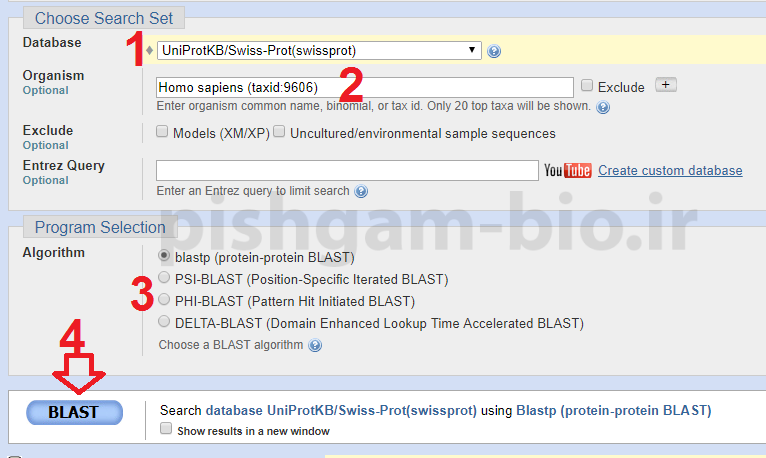
در این صفحه:
1: در قسمت Database از بین پایگاههای داده مختلف یک پایگاه را انتخاب نمایید
2: در قسمت Organism اسم یک گونه را وارد نمایید تا جستجو در توالیهای پروتئینی آن گونه انجام شود. در صورتی که هیچ گونه را وارد ننمایید از توالی پروتئینی همه موجودات برای بلاست استفاده میشود.
3: در قسمت Algorithm گزینه blastp (protein-protein BLAST) را انتخاب نمایید
و سپس بر روی گزینه Blast کلیک نمایید.
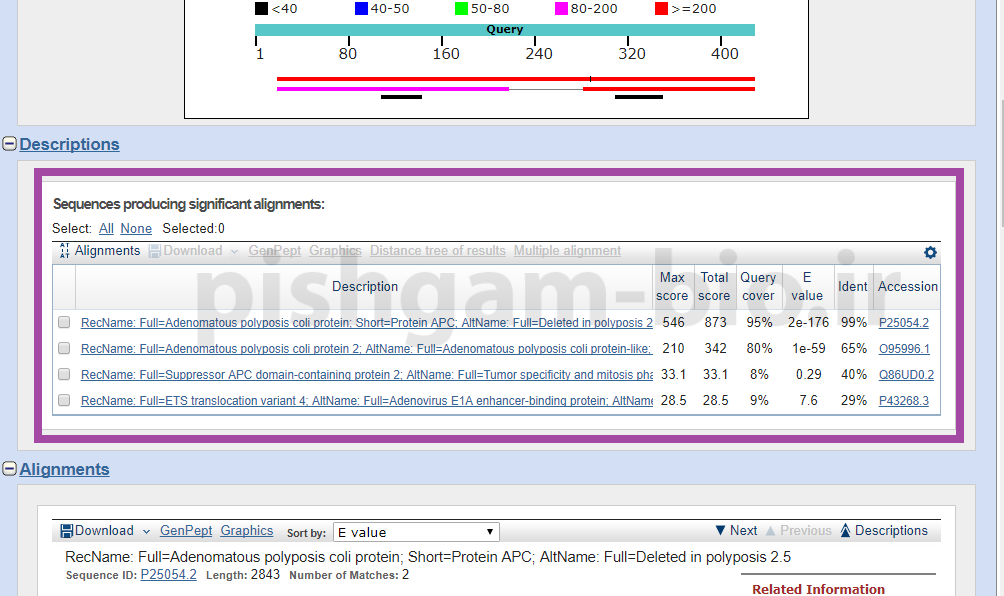
در صفحه نتایج بلاست و در قسمت Sequences producing significant alignments: میتوان ژنهایی که با توالی موردنظر شباهت دارند مشاهده نمود.
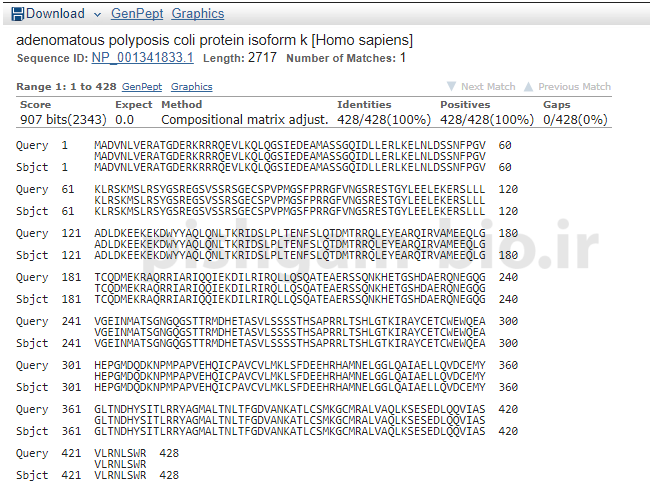
و در این قسمت میتوان هم ردیفی توالی پروتئینی مورد بررسی را با توالی پروتئین ای که مشابه آن میباشد مشاهده نمود.













دیدگاهتان را بنویسید