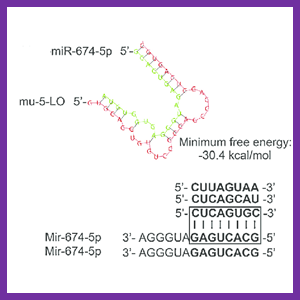
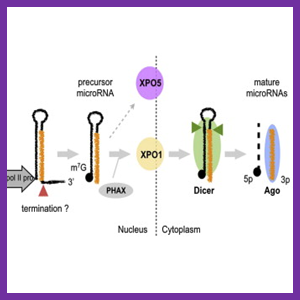
روش RNAi یک ابزار قدرتمند است که فقط از چند مولکول RNA دو رشته ای کوچک (dsRNA) برای خاموشی بیان ژن در سلول استفاده می کند و به عنوان روشی آسان و سریع برای تعیین عملکرد ژن در نظر گرفته شده است. این پدیده یک فرآیند مجاورتی طبیعی و خصوصیت درونی هر سلول در یک موجود چند سلولی است. گزارش ها نشان می دهد که روش RNAi ۱۰۰۰ برابر موثرتر از روش آنتی سنس در مطالعات خاموشی ژن و تعیین عملکرد آن می باشد بطوری که تنها 40 درصد از گیاهان تراریخته با استفاده از روش های آنتی سنس و سنس تولید شده اند.
آشنایی با مکانیسم روش RNA مداخله گر یا RNAi
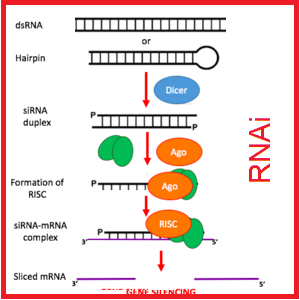
مکانیسم RNAi شامل دو مرحله اصلی است. در مرحله اول RNAهای دو رشته ای (dsRNA) به RNAهای مداخله کننده کوچک (siRNA) با طول ۲۳ – ۲۱ نوکلئوتید شکسته می شوند. این کار توسط آنزیم دایسر انجام می شود.
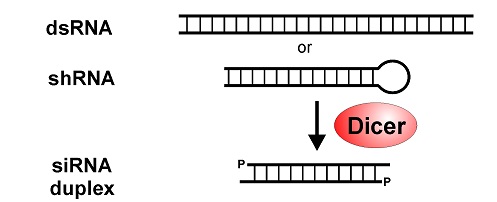
در حقیقت آنزیم دایسر، dsRNA را به siRNA دو رشته ای ۲۱ – ۲۰ نوکلئوتید فرآوری می کند. این مولکول ها دارای انتهای ۳ هیدروکسیل با دو نوکلئوتید اضافی آویزان و انتهای 5 پریم فسفوريله می باشند. ثابت شده که این ساختار siRNA ها برای مراحل بعدی RNAi ضروری می باشد. برای مثال دستکاری انتهای ه رشته آنتی سنس، مانع فعالیت siRNA می شود و انتهای صاف siRNA برای مراحل بعدی کارایی ندارد.
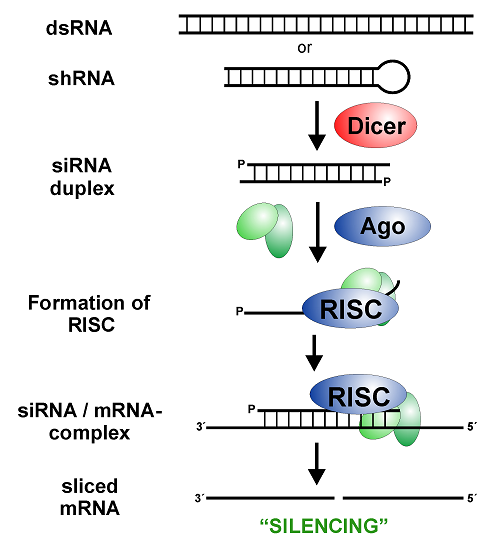
SiRNA های تولید شده بوسیله دایسر، به کمپلکس نوکلئازی RISC متصل می شوند که فرم بالقوه حاوی siRNA آن، برای تبدیل به فرم فعال، باید یک فرآیند را برای باز کردن دو رشته siRNA بوسیله یک هلیکاز (RNA هلیکاز وابسته به ATP) طی نماید که با مصرف انرژی همراه است. RISC همچنین حاوی یک اندوریبونوکلئاز است که با استفاده از توالی کد شده بوسیله رشته آنتی سنس siRNA، توالی مکمل در mRNA را یافته و تخریب می نماید. پروتئین ارگونات دارای دو دمین مشخص می باشد، PAZ (که در دایسر نیز یافت می شود) و PIWI.
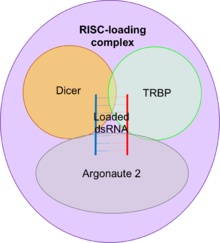
دمین PAZ بعنوان یک مدل متصل شونده به RNA برای آرگوناتها محسوب می شود که دو نوکلئوتید آویزان انتهای ۳ در siRNA ها را تشخیص می دهد و به آنها متصل می شود. تشخیص انتهای ۳ توسط PAZ موجود در دو خانواده پروتئینی، دایسرها و آرگوناتها برای تشخیص siRNA ها از سایر مولکولهای RNA اهمیت دارد. mRNA هدف به قطعات حدود ۲۲ نوکلئوتیدی (بین ۲۳-۲۱ نوکلئوتید) بریده می شود. وقتی برش کامل شد، RISC جدا شده و siRNA برای چرخه جدید تشخیص و برش mRNA مورد استفاده قرار می گیرد.
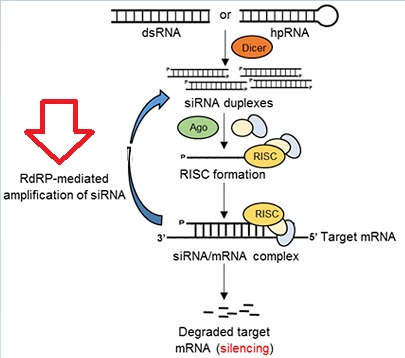
در گیاهان، آنزیم RNA پلیمراز وابسته به RNA (یا RdRp) یافت شده که مسئول تولید و تکثیر siRNA به dsRNA می باشد. این siRNA ها به عنوان آغازگرهایی برای تولید dsRNAهای جدید توسط RdRp بکار گرفته می شوند که متعاقبا این dsRNAها توسط آنزیم دایسر به siRNA های جدید فرآوری می گردند. در مجموع تجزیه mRNA هدف، 6-4 ساعت پس از ورود siRNA شروع می شود. بایستی siRNA با mRNA هدف بطور کامل جفت شود. این امر نیازمند اختصاصی بودن بسیار زیاد توالی های مکمل است.
تعدادی از آزمایش ها نشان می دهند که اگر توالی siRNA در یک باز با mRNA متفاوت باشد، کارایی siRNA برای جلوگیری از بیان ژن کاهش خواهد یافت و در صورتی که این تفاوت در ۳-۲ باز مشاهده شود، کارایی siRNA بطور کامل از بین خواهد رفت. تلاش های زیادی در زیست شناسی برای طراحی dsRNAهایی انجام شده است که حداکثر کاهش بیان با کمترین اثرات غیر هدف را دارا باشند. اثرات غير هدف هنگامی که توالی نوکلئوتیدی RNA قابلیت جفت شدن با چندین ژن را در یک زمان داشته باشد، مشاهده می شود. به عبارتی اختصاصی بودن کم توالى dsRNA با توالی ژن هدف منجر به بروز اثرات غیر هدف می شود. چنین مشکلاتی هنگامی که dsRNAها حاوی توالی های تکراری باشند، بیشتر واقع می شود.