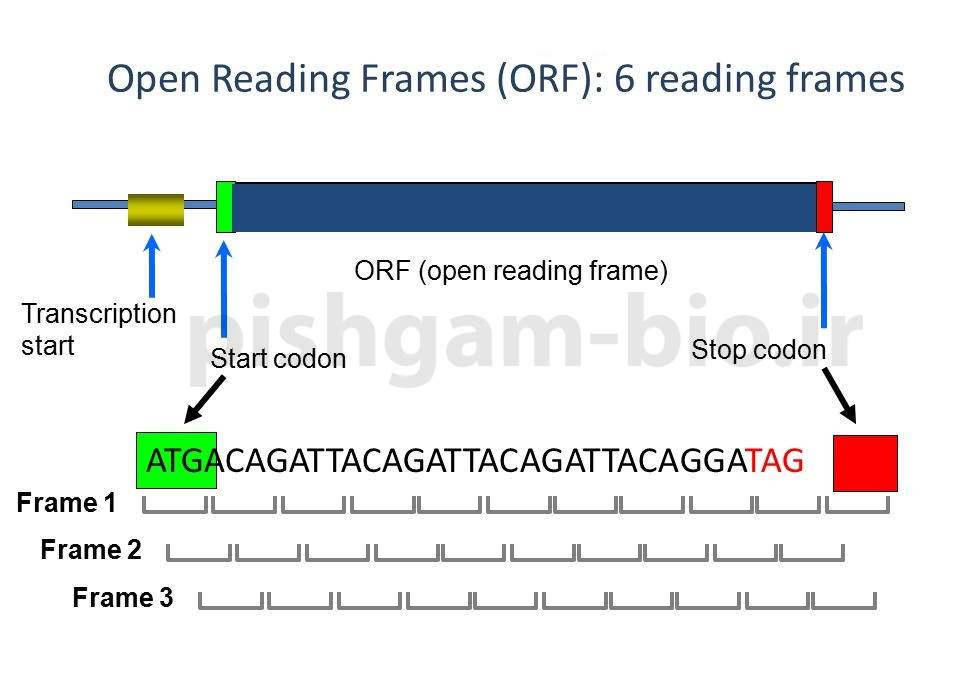
تعریف چهارچوب خوانش باز ( ORF ) چیست؟
در علم ژنتیک ، چهارچوب خوانش باز یا open reading frame (ORF)، به بخشی از توالی mRNA گفته میشود که قابل ترجمه به پروتئین باشد به عبارتی توالی ای که از خوانش کدون ها آن پروتئین سنتز می شود. ORF ، یک توالی به هم پیوسته از کدون های ژنتیکی می باشد که از کدون آغازین تا کدون خاتمه دهنده قرار گرفته است.
کاربرد شناسایی چهارچوب خوانش باز (ORF) در علم ژنتیک
یکی از کاربردهای متداول چهارچوب خوانش باز کمک به پیش بینی ژن است.
چهارچوب های خوانش باز بلند، اغلب به همراه سایر شواهد برای شناسایی نواحی منتخب کدکننده پروتئین یا نواحی کدکننده ی RNA فعال در توالی DNA به کار می روند.
یکی دیگر از کاربرد های بررسی نواحی ORF شناسایی پروتئین های کد شده توسط یک توالی و کشف عملکرد توالی های نا شناخته می باشد.
در ادامه آموزش نرم افزار translate از سایت Expasy برای شناسایی نواحی ORF توالی ها ارائه خواهد شد. برای یافتن نواحی ORF در نرم افزار translate می توان از توالی RNA و یا DNA استفاده نمود.
با کلیک بر روی لینک زیر به صفحه اصلی نرم افزار translate وارد می شوید.
https://web.expasy.org/translate/
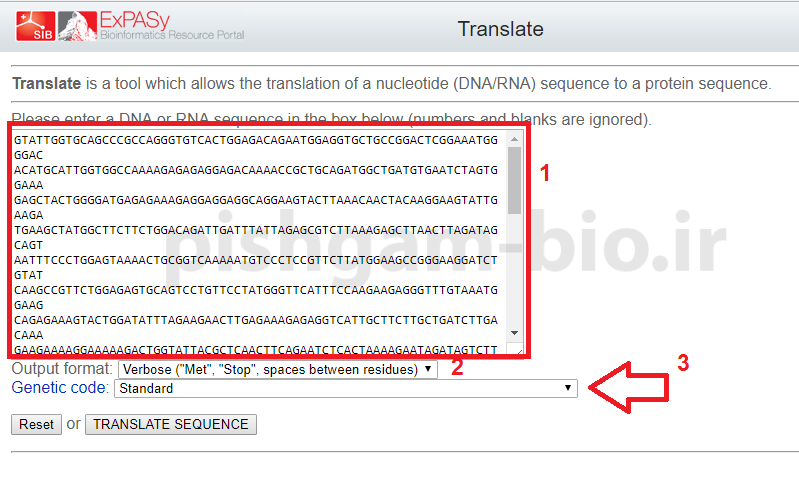
در پنجره نرم افزار translate ابتدا باید توالی مربوطه را در کارد مشخص شده وارد نمایید.
سپس در قسمت Output format تعیین نمایید که نتایج به چه صورتی نمایش داده شوند، البته حالت پیش فرض نرم افزار بهترین گزینه می باشد.
در مرحله سوم و در قسمت Genetic code باید تعیین نمایید که از چه کد ژنتیکی برای شناسایی ناحیه ORF استفاده شود.
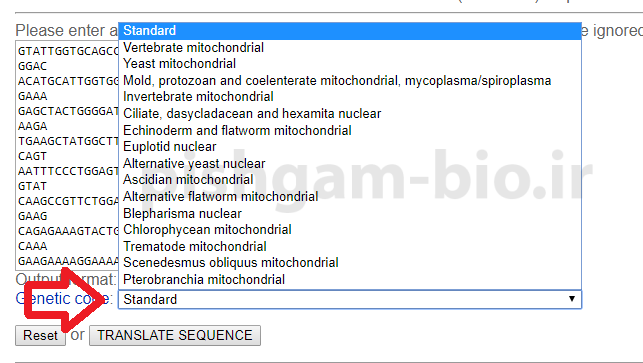
از آن جایی که کد ژنتیکی در برخی از موجودات مانند مخمر و حتی میتوکندری مهرداران با کد ژنتیکی استاندارد متفاوت می باشد باید در قسمت Genetic code مشخص نمایید با چه کد ژنتیکی وجود نواحی ORF بررسی شود. برای ژنوم انسان و بیشتر موجودات می توان از کد ژنتیکی standard استفاده نمود.
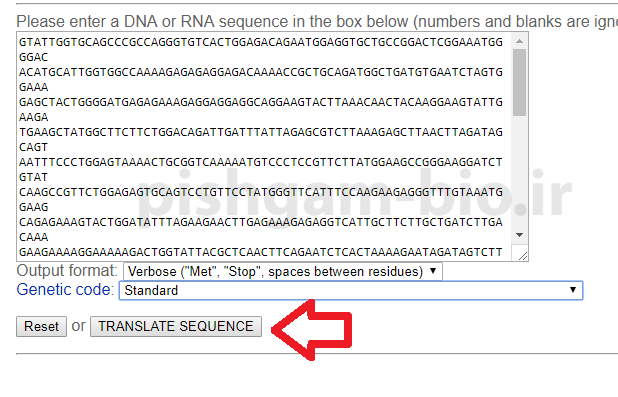
بعد وارد نمودن توالی و انتخاب نوع کد ژنتیکی بر روی گزینه Translate sequence کلیک نمایید تا توالی بررسی شود و نتایج در صفحه بعدی نمایش داده شود.

نرم افزار Translate وجود ORF را در 6 قالب ممکن بررسی می نمایید، از آن جایی که هر کدن ژنتیکی از 3 باز تشکیل شده است برای هر توالی می توان 6 قالب خوانش در نظر گرفت که سه قالب روی رشته + (همان رشته ای که وارد نموده اید) و 3 قالب روی روی رشته – ( توالی reverse complement رشته وارد شده) قرار گرفته است.
برای شناسایی ORF، نرم افزار ابتدا آسیدآمینه met (متیونین) را که اولین کدن ORFها می باشد شناسایی می نماید (زیرا فرایند ترجمه پروتئین از اسیدآمینه متیونین آغاز میشود) و سپس کدن های توالی را تا رسیدن به یک کدن پایان ادامه می دهد سپس توالی مربوطه را به عنوان ORF معرفی می نماید.
شکل فوق نتایج وجود ORF را در سه قالب رشته + نشان می دهد که با علامت 5’3′ Frame نمایش داده می شوند . و همانگونه که قابل مشاهده می باشد قالب 5’3′ Frame2 دارای ORF با بیشترین طول می باشد

در شکل فوق نتایج وجود ORF را در سه قالب رشته – نشان می دهد که با علامت 3’5′ Frame نمایش داده می شوند.
با بررسی 6 قالب، به احتمال فروان قالب 5’3′ Frame2 باید ORF توالی وارد شده باشد. اما باید توجه نمود که وجود ORF با طول زیاد بیشتر در توالی mRNA مشاهده میشود اما اگر توالی ژنومی مورد بررسی قرار گیرد به دلیل وجود اینترون های بزرگ در توالی یوکاریوت ها احتمال کمی برای مشاهده ORF طولانی وجود دارد.














دیدگاهتان را بنویسید