معرفی نرم افزار Ntsys
نرم افزار NTsys یک نرم افزار بسیار قدرتمند جهت انجام چندین آزمون آماری چند متغیره نظیر تجزیه به مؤلفه های اصلی، Correspondence analysis، تجزیه همبستگی های کانونی و چند آزمون آماری چند متغیره دیگر می باشد با این وجود مهمترین بخش این نرم افزار تجزیه خوشه ای یا Cluster analysis است که بطور منحصر به فردی گسترش یافته است. در این نرم افزار شما می توانید با استفاده از انواع ضرایب فاصله، دندروگرام (درخت تجزیه) ژنوتیپ های مورد آزمون را با روش های مختلف تجزیه خوشه ای نظیر UPGMAT،CL و غیره رسم کنید. همچنین نمودار رسم شده کاملاً قابل ویرایش است.
مراحل ترسیم درخت فیلوزنتیکی توسط نرم افزار NTsys
- انتخاب مارکر مناسب که توانایی تفکیک افراد و جمعیت ها را از هم داشته باشد مانند مارکر های SSR, ISSR, RAPD و یا سایر مارکرهای سیستماتیک
- ایجاد یک فایل وروری برای داده های حاصل از مارکرهای فوق برای نرم افزار NTsys
- محاسبه ضرایب تشابه و تشکیل ماتریس شباهت بین افراد
- ادغام و گروهبندی افراد مانند UPGMA، NJ
- بررسی کارایی روش گروه بندی
- بررسی افراد با دسته بندی PCO
- بررسی تشابه و همخوانی دو درخت فیلوزنتیکی که با دو مارکر مختلف حاصل شده اند
- ادغام درخچه ها های مختلف و ترسیم درخچه consensus
نحوه مرتب سازی داده ها و ایجاد یک فایل وروری برای داده های حاصل از مارکرهای فوق برای نرم افزار NTsys
برای شروع یک مثال ساده را توضیح میدهیم
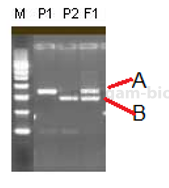
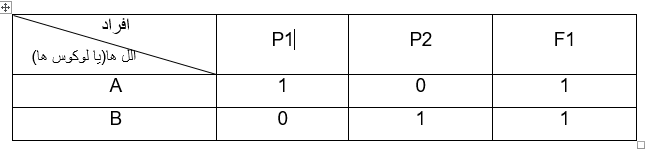
در این شکل ما یک مارکر SSR را بررسی نموده ایم که داری دو آلل است و فرد مورد بررسی نیز دیپلوتایپ می باشد و در این بررسی سه فرد وجود دارد که عبارتند از P1, P2 و F1 . حال می خواهیم داده های این سه نفر را در جدول وارد نماییم که بعدا از آن جهت خوشه بندی افراد استفاده نماییم. به صورت توافقی آلل بالا را A والل پایین را B می نامیم برای حضور باند (الل) عدد 1 و عدم حضور باند عدد 0 را اختصاص می دهیم پس ژنوتیپ افراد به این صورت می شود:
حال با نحوه امتیاز دهی به افراد اشنا شدید، تعداد افراد و مارکر های مورد بررسی را بیشتر مینماییم 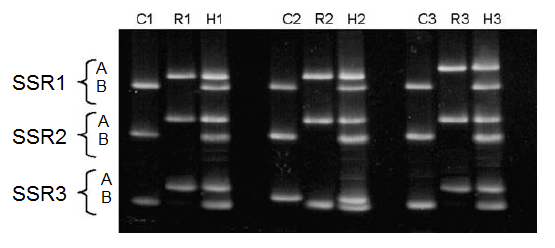
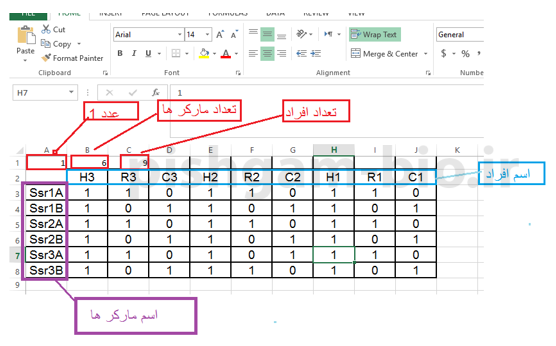
در این شکل ما با سه مارکر SSR( شامل لوکوس های SSR1, SSR2, SSR3) سه جمعیت را مورد بررسی قرار داده ایم که هر جمعیت شامل 3 فرد است. پس در مجموع 9 فرد در سه جمعیت داریم. برای شکل فوق میخواهیم یه فایل ورودی داده برای نرم افزار NTsys درست نماییم.
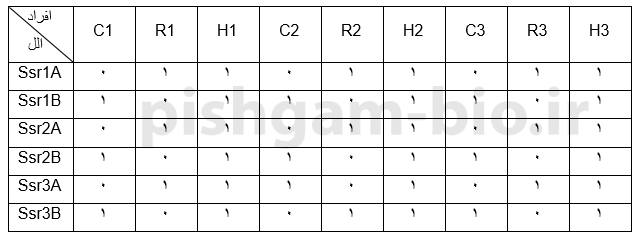
در ابتدا مشخص نماییم هر الل را چه بنامیم. می توان در هر لوکوس به صورت توافقی الل بالا را A و الل پایین را B بنامیم و برای هر سه لوکوس از علامت های A وB استفاده نماییم. پس ژنوتیپ افراد به این صورت جدول زیر می شود.
آموزش وارد نمودن داده ها به نرم افزار NTsys
برای وارد نمودن داده ها در نرم افزار NTsys دو روش وجود دارد. اول وارد کردن دستی داده در نرم افزار که این حالت برای تعداد نمونه کم کاربرد دارد. دوم تنظیم یک فایل اکسل برای وارد نمودن داده ها و سپس import داده ها به نرم افزار NTsys
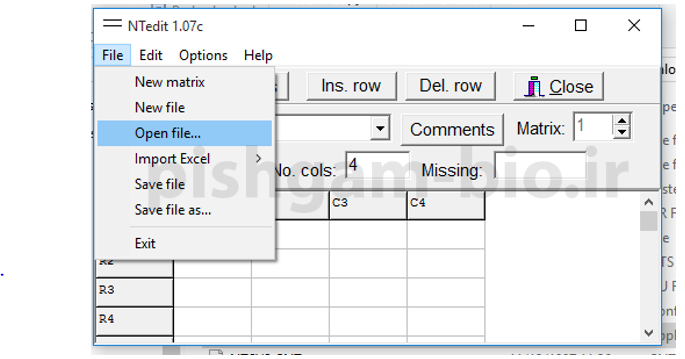
روش مستقیم وارد نمودن داده ها به نرم افزار ntsys ابتدا نرم افزار ntedite را باز نمایید.
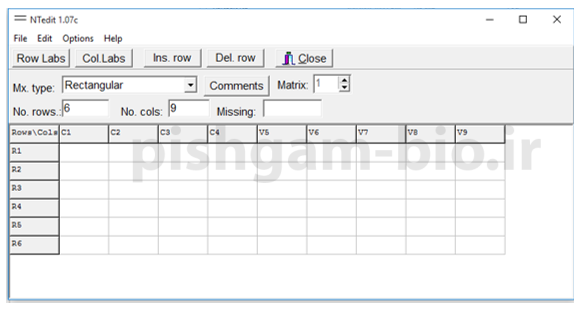
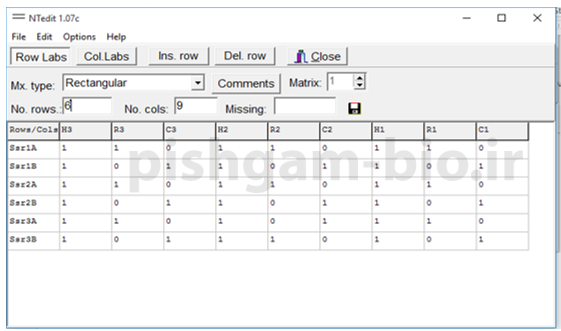
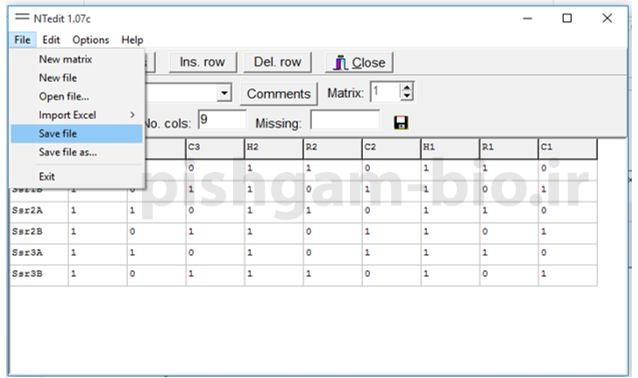
برای تعداد ردیف ها تعداد مارکرها را وارد نمایید ( در جدول قبلی 6 لوکوس وجود داشت بنابراین اینجا هم عدد 6 را وارد میکنیم) و در n. cols: عدد 9 که برابر تعداد افراد مورد بررسی است را وارد می نماییم و سپس برروی Row lable کلیک کنید و اسم ردیف ها مطابق با اسم مارکرها تعیین کنید و سپس برروی col. lable کلیک کنید و اسم ستون ها مطابق با اسم مارکرها تعیین کنید و سپس جدول را امتیاز هر فرد برای هر مارکر مانند شکل های زیر پر نمایید و سپس آن را دخیره نمایید.
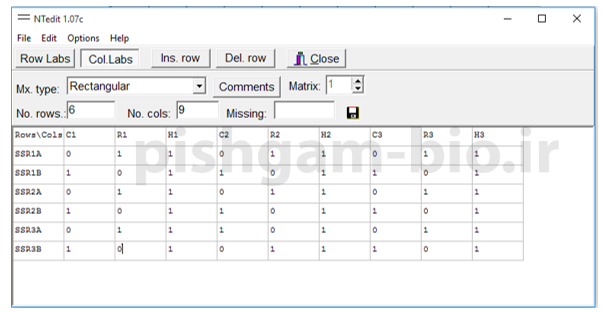
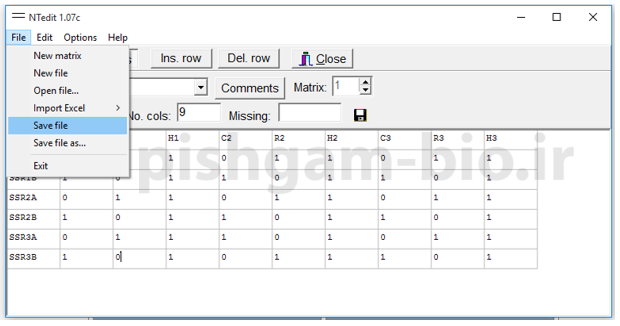
بعد از دخیره کردن داده های وارد شده، فایل دخیره شده را به عنوان یک فایل ورودی خام جهت بررسی های فیلوژنتیکی استفاده می نماییم.
برای مواقعی که تعداد افراد و تعداد مارکرها زیاد باشد می توان از طریق یک فایل اکسل داده ها را آماده نمود. فایل اکسل را به شکل زیر آماده نمایید و حتما توجه نمایید که در اولین خانه (A1) عدد 1 را قرار بدهید. و در نهایت فایل اکسل را به فرمت 2003 (.xls) دخیره نمایید.
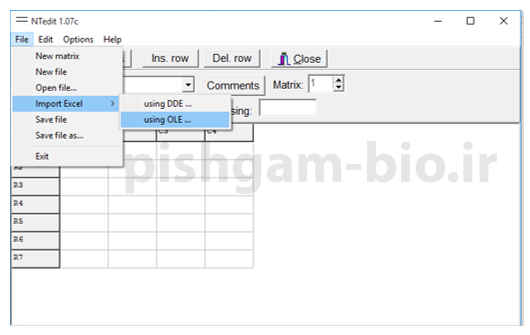
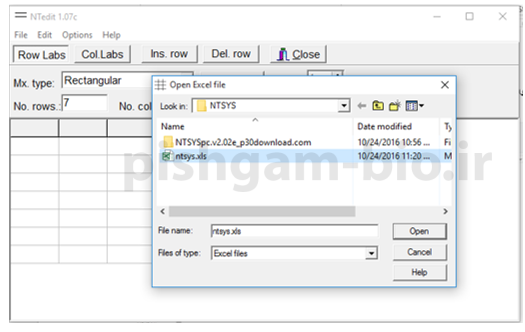
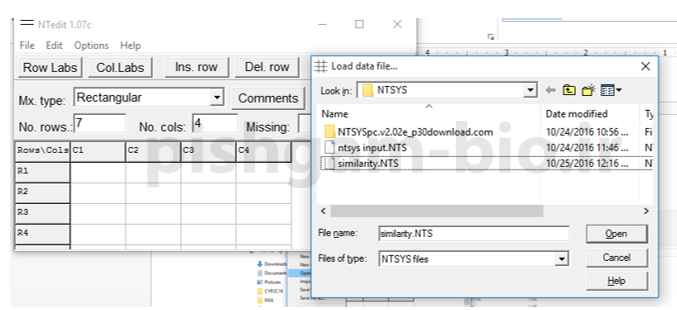
برای وارد نمودن داده های فوق در نرم افزار ntedit طبق عکس های زیر عمل نمایید و سپس فایل نهایی را با نام ntsys input دخیره نمایید.
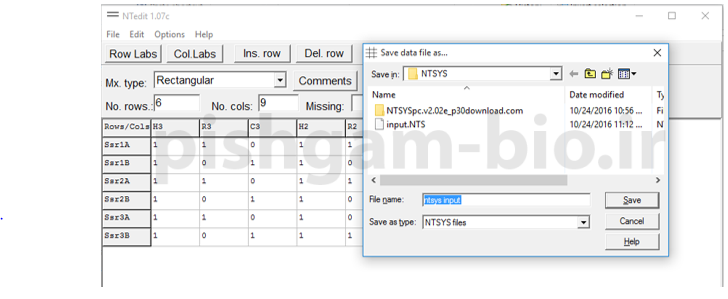
بعد از تهیه فایل فوق می توان داده ها را توسط نرم افزار NTsys بررسی نمود.
آموزش آنالیز داده ها با نرم افزار NTsys و محاسبه ضرایب فاصله ای و تشابه مانند Dice ، jacard ، SM

مرحله اول در دسته افراد بررسی ضرایب تشابه بین آنها می باشد. برای بررسی ضرایب تشابه به زبانه similarity بروید در این زبانه می توانید با انتخاب دو گزینه simlnt و simqual ضرایب تشابه را برای افراد محاسبه نمود. هر کدام از این دو گزینه دارای روش های خاصی برای محاسبه ضرایب تشابه را برای افراد می باشند که در شکل های زیر به آن ها اشاره شده است. برای محاسبه ماتریس ضرایب تشابه بعد از انتخاب دو گزینه فوق مانند شکل های زیر عمل کنید.
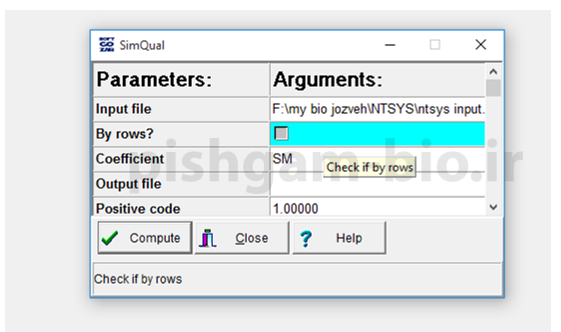
در ابتدا به دلیل اینکه مارکرهای ما در ردیف ها قرار گفته اند ( در ساختن قایل اولیه و امتیازدهی) تیک گزینه by row? را نمی زنیم تا بررسی براساس ستونها که افراد می باشند انجام گیرد(اگر این کار را کنید در اخر درخت فیلوژنتیکی برای مارکرها ترسیم میگردد نه افراد).
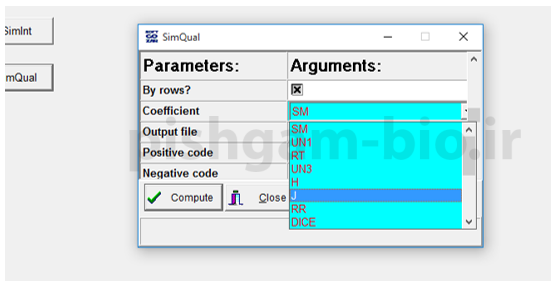
سپس ضریب دلخواه خود را انتخاب نمایید ( برای انتخاب ضریب مناسب از مقالات کمک بگیرید).
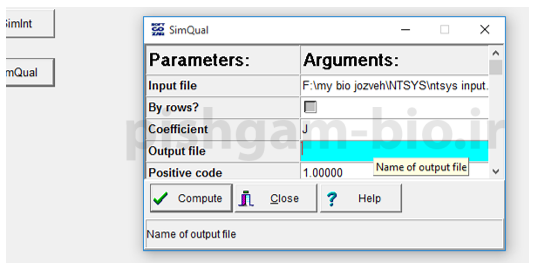
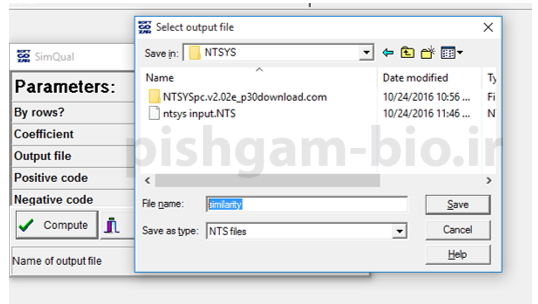
سپس با دوبار کلیک کردن برروی گزینه output file یک مسیر و اسم را برای ذخیره فایل حاوی ماتریس تشابه افراد انتخاب کنید.
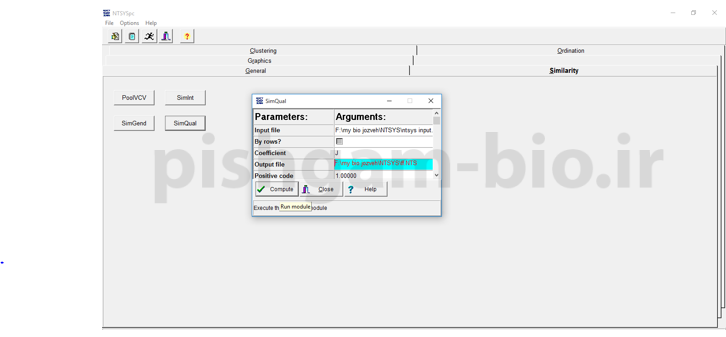
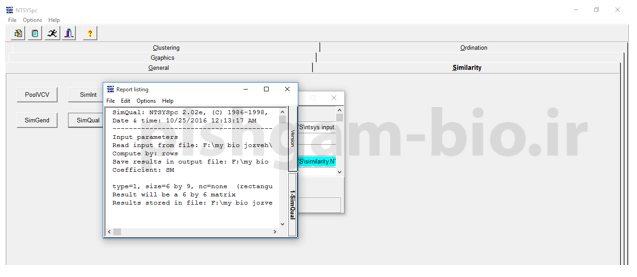
بعد از کلیک برروی گزینه compute و باز شدن پنجره فوق بیانگر اتمام محاسبه ضرایب می باشد. به فولدری که تعیین کرده بودید بروید فایل ضرایب تشابه در آنجا ذخیره شده است. که می توانید آن را با نرم افزا ntedit باز و ضرایب را مشاهده فرمایید.
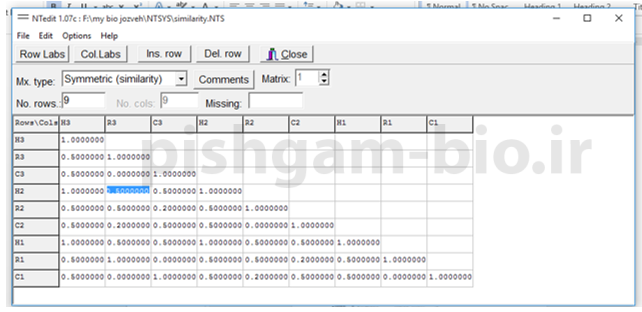
در شکل فوق ضرایب تشابه را بین افراد مشاهده می فرمایید.
برای ترسیم درخت فیلوژنتیکی از زبانه clustering استفاده میکنیم
آموزش ترسیم درختچه فیلوژنتیکی و clustering مانند neighbor joining، UPGMA، WPGMA با نرم افزار Ntsys pc
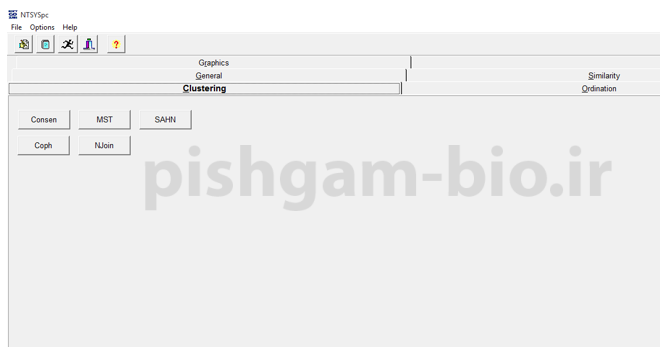
برای ترسیم درخت neighbor joining از گزینه NJion استفاده میکنیم سایر روش های ترسیم درخت نیز در گزینه SAHN قرار دارند.
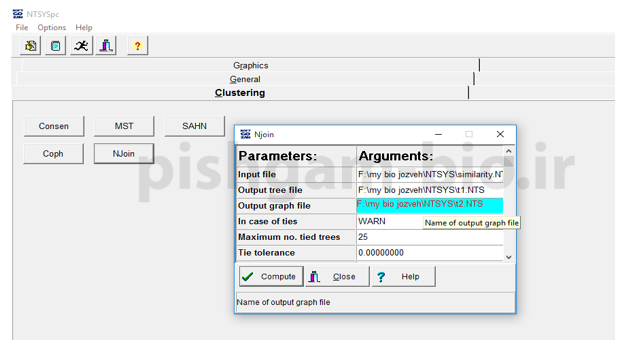
در ترسیم درخت neighbor joining بعد از انتخاب گزینه NJion برای input file همان فایل ضرایب تشابه را وارد نموده و سپس در گزینه های output tree file و output graph file دو اسم برای فایل های خروجی انتخاب میکنیم و سپس برروی compute کلیک میکنیم.
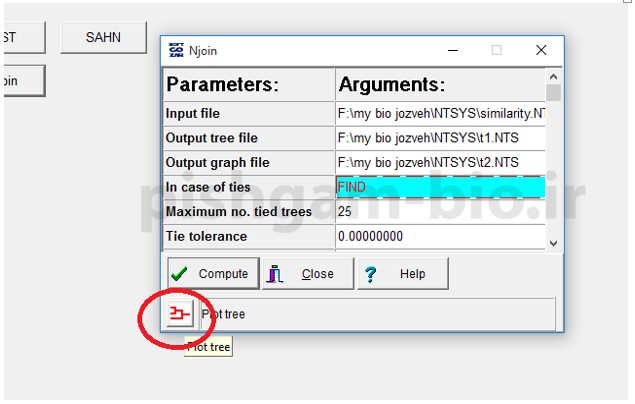
در نهایت با کلیک برروی گزینه مشخص شده در شکل فوق می توانید درخت ترسیم شده را مشاهده نمایید. همچین دو فایل خروجی نیز در پوشه تعیین شده برای درخت ها ایجاد شده که به کاربرد آن ها در ادامه اموزش اشاره میگردد.
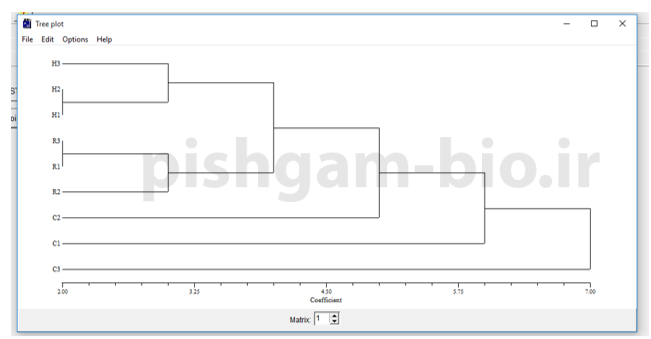
درخت ترسیم شده به روش neighbor joining
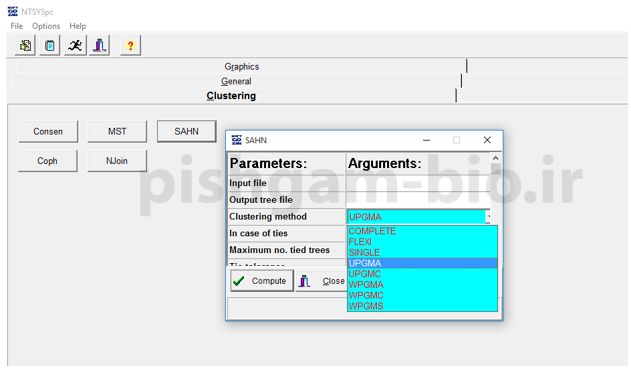
شکل فوق: ترسیم سایر انواع درخت ها نیز شبیه روش neighbor joining می باشد ولی باید در گزینه clustering method نوع درخت را انتخاب نمود.
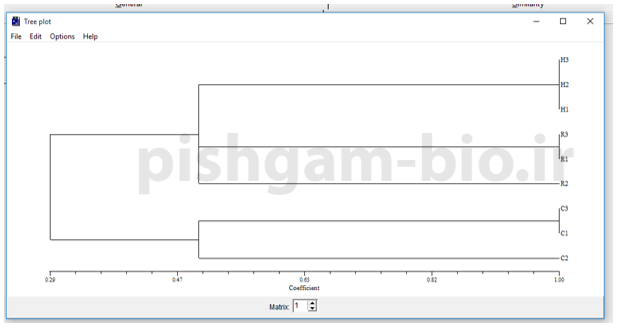
درخت ترسیم شده به روش UPGMA
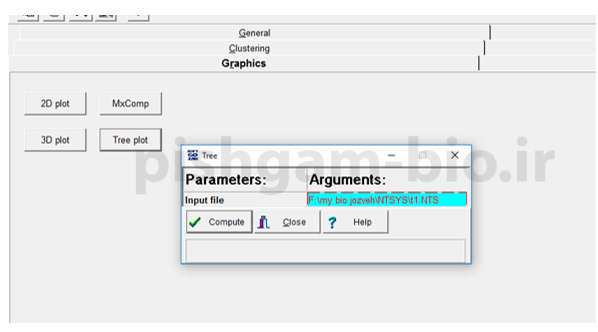
شکل فوق: در صورتی که بخواهید درخت ترسیم شده را دوباره مشاهده فرمایید به زبانه graphic بروید و گزینه tree plot را انتخاب نمایید سپس فایل ذخیره شده درخت را به آن معرفی نمایید و compute را کلیک نمایید تا درخت ترسیم شده باز شود.
تعیین صحت درخت فیلوژنتیک ترسیم شده با ضریب کوفنتیک (cophenetic) با نرم افزار ntsys pc
در شکل های بالا مشاهده نمودید که دو درخت ترسیم شده با روش های مختلف باهم تفاوت دارند حال کدام درخت صحیح تر میباشد؟
ضریب کوفنتیک چیست
برای تعیین صحت درخت ترسیم شده از ضریب کوفنتیک استفاده میگردد هر چه این ضریب به 1 نزدیک باشد اعتبار درخت بیشتر می باشد.
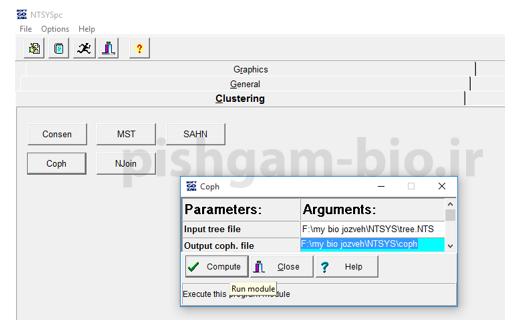
برای محاسبه ضریب کوفنتیک از زبانه clustering گزینه coph را انتخاب نمایید و سپس فایلی که در مراحل قبل برای درخت ذخیره شده بود به نرم افزار معرفی نمایید و سپس یک فایل خروجی برای نتایج تعیین کنید و compute را کلیک کنید بعد از محاسبه یک قایل ذخیره شده در فولدر تعیین شده مشاهده خواهید کرد که برای تعیین ضریب کوفنتیک از آن استفاده میشود در واقع در این فایل جدید ضرایب تشابه بین افراد براساس درختچه محاسبه شده است. بنابراین با مقایسه و تعیین میزان تفاوت ان با ضرایب تشابه واقعی میتوان اعتبار درخت را تعیین کرد.
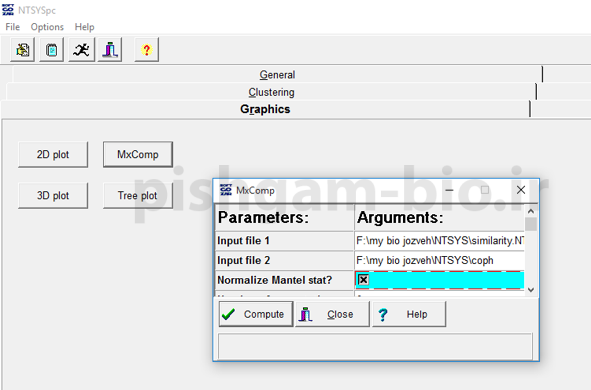
برای تعیین ضریب کوفنتیک به زبانه graphics بروید و گزینه mxcomp را انتخاب نمایید. در پنجره باز شده ضرایب تشابه افراد واقعی و ضرایب تشابه محاسبه شده بر اساس درخت را به عنوان input file شماره 1 و 2 وارد نمایید و در بخش Number of permutations عددی بین 1 تا 999 را وارد نمایید (معمولا عدد900 را وارد میکنند. به این مطور که دو ماتریس 900 بار با هم مقایسه شوند) و سپس compute را کلیک کنید.
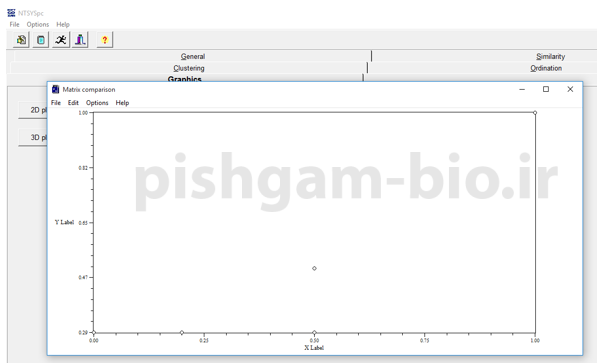
این نمودار هر چه به خط y=x منطبق تر باشد ضریب کوفنتیک بالاتر می باشد.( چون تعداد مارکرها و نمونه های ما بسیار کم و 6 عدد بود نمودار به زیبایی رسم نشده است).
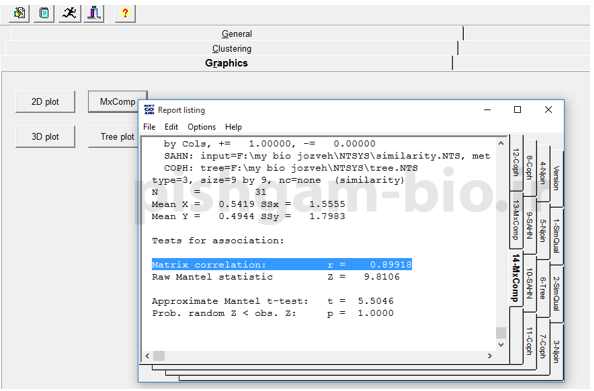
در report listing که بعد از هر محاسبه باز میشود مقدار matrix correlation را پیدا نموده، این عدد همان ضریب کوفنتیک می باشد.
مقایسه دو درخت های فیلوژنتیک با یکدیگر با استفاده از آزمون مانتل و ضریب کوفنتیک
فرض نمایید با استفاده از مارکرهای مولکولی ISSR و RAPD افراد یکسانی را خوشه بندی نموده ایم حال میخواهیم ببینیم این دو درحت چه قدر با هم همخوانی دارند. برای این کار از ازمون مانتل ( همان ضریب کوفنتیک ) استفاده میشود. در این حالت میتوان ضرایب تشابه محاسبه شده ( یا ضرایب تشابهی که از طریق درخت ها محاسبه شده اند) توسظ دو مارکر را با هم مقایسه نمود و ضریب کوفنتیک را محاسبه نمود برای این کار همانند روش های فوق عمل نمایید.
نحوه ترسیم درخت فیلوژنتیک حفاظت شده consensus با نرم افزار Ntsys pc
فرض نمایید با استفاده از مارکرهای مولکولی ISSR و RAPD افراد یکسانی را خوشه بندی نموده ایم حال میخواهیم با ترکیب این دو درخت یه درخت مورد توافق از هر دو مارکربه دست بیاوریم. برای این کار به زبانه clustering رفته و گزینه consen را انتخاب مینماییم. برای فایل های input شماره 1 و 2 فایل ذخیره شده برای درخت برای هر دو مارکر تعیین میگردند و سپس یک فایل خروجی برای ترسیم درخت مورد توافق در نظر بگیرید.
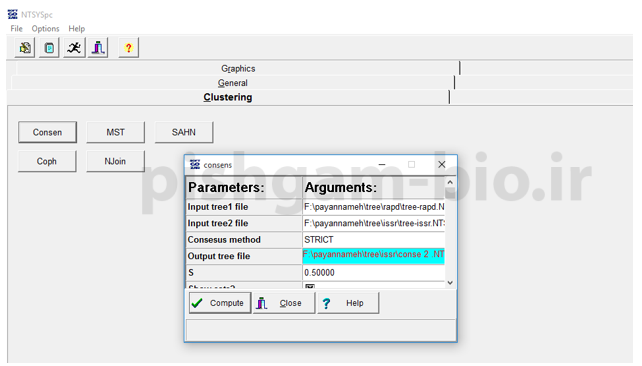
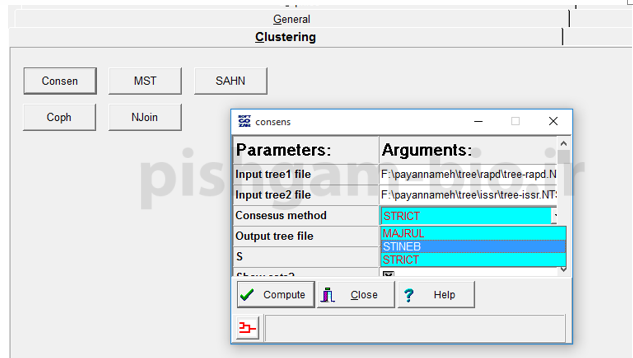
شما در بخش consensus method سه گزینه مشاهده میفرمایید که گزینه strict سخت گیرانه ترین روش برای ترسیم درخت مورد توافق می باشد.
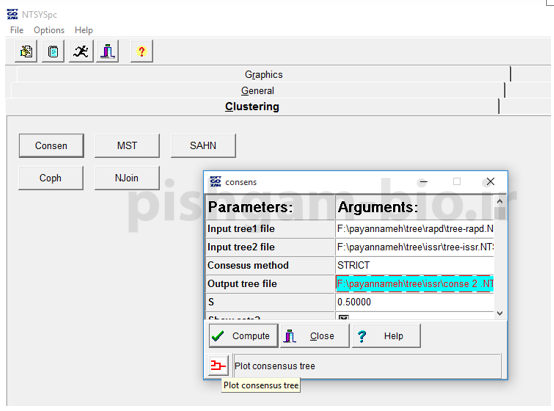
با کلیک برروی این گزینه (گوشه پایین سمت راست) می توانید تصویر درخت ترسیم شده را ملاحظه بفرمایید.
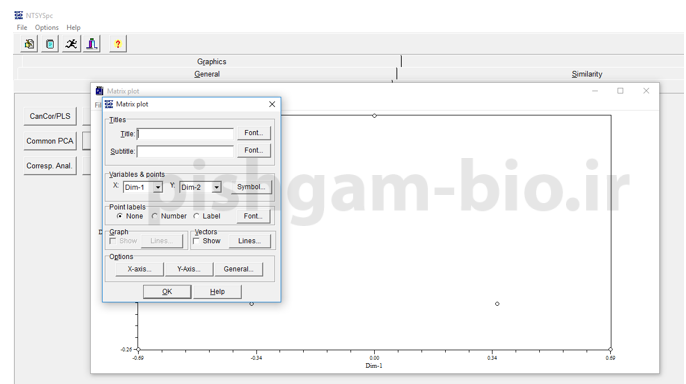
آموزش آزمون تجزیه به مولفه های اصلی (PCA) دو بعدی و سه بعدی با استفاده از نرم افزار Ntsys pc
برای انجام ازمون تجزیه به مولفه های اصلی ابتدا به زبانه general رفته و سپس گزینه Dcenter را انتخاب می کنیم (شکل زیر)
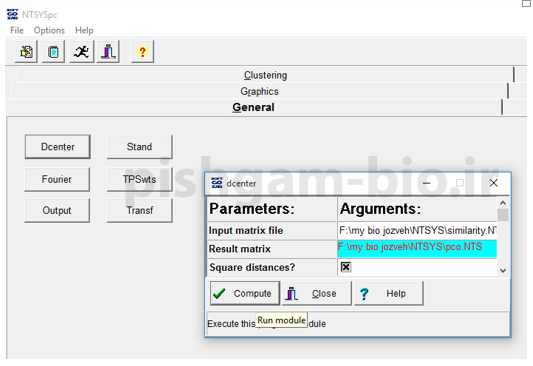
برای input matrix file فایل مربوط به ماتریس ضرایب تشابه افراد را معرفی نموده و سپس برای فایل داده های خروجی یک مسیر انتخاب میکنیم. که بعدا از این فایل جهت ازمون تجزیه به مولفه های اصلی استفاده خواهیم نمود. برای این که به زبانه ordination می رویم و گزینه Eigen را انتخاب میکنیم ( شکل زیر)

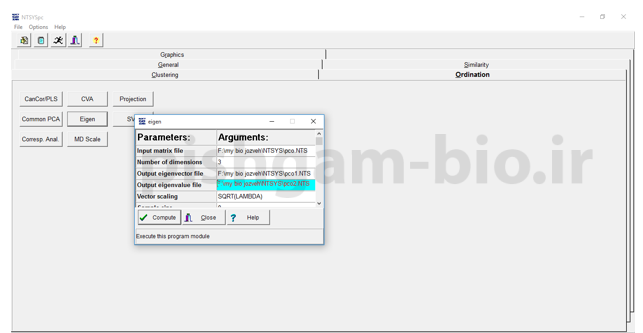
برای فایل input همان فایلی که در قسمت بالا (زبانه general و سپس گزینه Dcenter) را معرفی مینماییم و سپس دو فایل نیز برای داده های خروجی تعیین میکنیم و سپس compute را کلیک میکنیم.
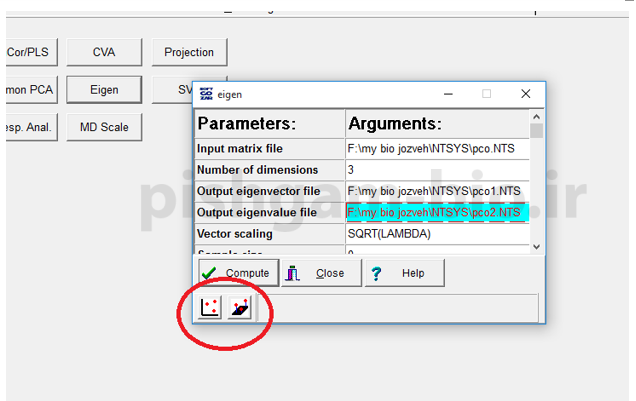
با کلیک برروی دو گزینه مشخص شده می توان نمودار های دو بعدی و سه بعدی ترسیم شده را ملاحظه نمود.
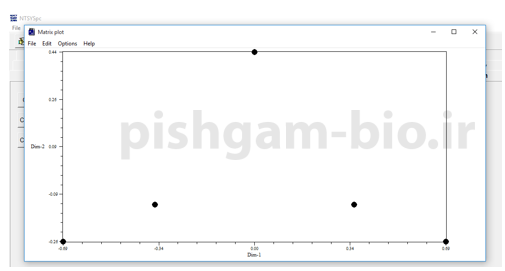
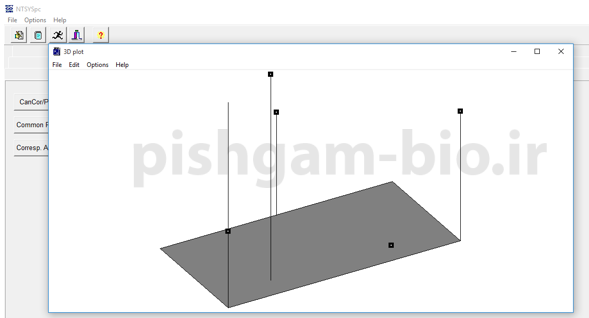
با استفاده از گزینه های زیر میتوانید شکل نمودار ترسیم شده را زیباتر نمایش بدهید.















دیدگاهتان را بنویسید